- Beiträge: 1748
Sidebar
- Forum
- PH-Forum
- Forschung und Wissen
- Aus mesenchymalen Stammzellen der menschlichen Nabelschnur gewonnene Behandlung
Aus mesenchymalen Stammzellen der menschlichen Nabelschnur gewonnene Behandlung
10 Jun 2022 16:25 #1501
von danny
OMNIA TEMPUS HABENT
Diagnose IPAH im Februar 2013, in Behandlung bei OA Dr. Ulrich Krüger, jetzt Dr. Fischer Herzzentrum Duisburg, Medikamente: Sildenafil, Bosentan jetzt Macitentan, Subkutane Treprostinilpumpe, seit Januar 2024 getunnelter ZVK mit externer Pumpe (Groshongkatheter), 24/7 Sauerstoff, Marcumar, Diuretika
Aus mesenchymalen Stammzellen der menschlichen Nabelschnur gewonnene Behandlung wurde erstellt von danny
Aus mesenchymalen Stammzellen der menschlichen Nabelschnur gewonnene Behandlung von schwerer pulmonaler arterieller Hypertonie
www.nature.com/articles/s44161-022-00083...gyI9sDcQAbstraktHier berichten wir über die Anwendung einer humanen Nabelschnur-Mesenchym-Stammzelle (HUCMSC)-abgeleitete Therapie für pulmonale arterielle Hypertonie (PAH). Eine 3-jährige Frau stellte sich mit vererbbarer PAH in Verbindung mit erblicher hämorrhagischer Teleangiektasie vor und wurde 6 Monate lang mit seriellen intravaskulären Infusionen von konditionierten Medien (CM) aus allogenen HUCMSCs behandelt. Die Behandlung verbesserte die klinischen und hämodynamischen Parameter deutlich und verringerte Blutplasmamarker für Gefäßfibrose, Verletzungen und Entzündungen. Eine vergleichende Analyse von Einzelzell-RNA-Sequenzierungsdaten, die von drei HUCMSCs und zwei humanen Nabelvenen-Endothelzellen (HUVEC)-Kontrollen gesammelt wurden, identifizierte acht gemeinsame Zellcluster, die alle auf ein für HUCMSCs spezifisches regeneratives Potenzial hindeuteten. Die Eigenschaften von HUCMSCs wurden durch nicht zielgerichtete, markierungsfreie Quantifizierung des Zell- und CM-Proteoms validiert, was auf eine erhöhte Aktivität der Regeneration, Autophagie und entzündungshemmender Wege und der mitochondrialen Funktion hindeutet. Die Prostaglandinanalyse zeigte eine erhöhte HUCMSC-Sekretion von Prostaglandin E2, das für seine Regenerationsfähigkeit bekannt ist. Zusätzliche prospektive klinische Studien sind gerechtfertigt, um die Vorteile einer von HUCMSC abgeleiteten Therapie für PAH zu bestätigen und weiter zu untersuchen.HauptsächlichPAH ist gekennzeichnet durch fortschreitenden, obliterativen Umbau der Lungenarteriolen, präkapillären Gefäßverlust, Rechtsherzinsuffizienz und Tod. Die Pathobiologie von Lungengefäßerkrankungen (PVD) und PAH ist komplex, multifaktoriell und wird durch Entzündungen und Stoffwechselstörungen vorangetrieben 1 . Trotz bemerkenswerter Verbesserungen in der Pharmakotherapie 2 , 3 ist fortgeschrittene PAH immer noch eine nicht heilbare, schwächende und tödliche Erkrankung 4 . Stamm- und Vorläuferzellen und/oder ihre sekretierten Produkte können effizientere Therapien für PAH darstellen 5 , 6 . Mitglieder der Transforming Growth Factor Beta (TGFβ)-Rezeptor-Superfamilie (Bone Morphogenetic Protein Receptor 2, BMPR2; Aktivin-A-Rezeptor-ähnlicher Typ 1, ACVRL1 ; und Endoglin, ENG ) und ihre Liganden spielen eine entscheidende Rolle in der Ätiologie von PAH 1 , 7 , 8 , 9 . Heterozygote Loss-of-Function-Mutationen in den Genen BMPR2 , ACVRL1 und ENG wurden unter anderem bei familiärer/heritabler PAH (HPAH) und idiopathischer PAH (IPAH) beschrieben 9 ; solche Mutationen finden sich auch bei hereditärer hämorrhagischer Teleangiektasie (HHT; Osler-Weber-Rendu-Krankheit). Patienten mit ACVRL1- Mutationen, die PAH entwickeln 7 sind besonders jung, haben oft einen raschen Krankheitsverlauf und eine schlechtere Prognose als Patienten mit BMPR2- Mutationen 10 . Hier demonstrieren wir eine sichere und effiziente HUCMSC-abgeleitete Behandlung schwerer, fortschreitender PAH mittels serieller intravaskulärer Infusionen von HUCMSC-CM bei einem jungen Patienten mit vererbbarer PAH und HHT Typ 2, verursacht durch eine ACVLR1 - Missense-Mutation.ErgebnisseZum Zeitpunkt der Diagnose befand sich das 3-jährige Mädchen in einem kritischen Zustand, nach zwei synkopalen „afebrilen Anfallsepisoden“, in Funktionsklasse 4 der Weltgesundheitsorganisation (WHO), mit einer 6-minütigen Gehstrecke (6MWD) von nur 270 Metern (SpO 2 > 95 %) und mäßige Thrombozytopenie bei 8 × 10 3 µl. Sie litt seit 10 Monaten unter Müdigkeit, wiederholtem Nasenbluten (Epistaxis) und mukokutanen Teleangiektasien an den Lippen, der Brust und den unteren Extremitäten. Serum-NTproBNP war mit 2.414 ng l –1 stark erhöht. Die Echokardiographie zeigte eine stark beeinträchtigte rechtsventrikuläre systolische Funktion (systolische Exkursion der Trikuspidalringebene (TAPSE), 1,4 cm) und Trikuspidalinsuffizienz, Grad 2. Die erste diagnostische Herzkatheterisierung (CATH #0; behandlungsnaiv) wurde im Januar 2019 an einem externen Tertiär durchgeführt Center. Invasive hämodynamische Messungen zeigten schwere suprasystemische PAH (Druck: Pulmonalarterie (PA), 119/57/85 mmHg; aufsteigende Aorta (AAO), 94/43/63 mmHg; mittlerer pulmonalarterieller Druck (mPAP)/mittlerer systemischer arterieller Druck (mSAP ) Ratio 1,35), stark erhöhter pulmonalvaskulärer Widerstand (PVRi, 21 WU × m²; PVR/SVR Ratio 1,2), Mangel an akuter Vasoreaktivität (AVT) und normaler Herzindex (Qsi, 3,6 l min −1 m −2). Dementsprechend erreichte der Patient bei der Diagnose eine „höhere Risiko“-Stratifizierung mit einem höheren Risiko-Score des European Pediatric PVD Network (EPPVDN) von bis zu 0,8 (12/15) und einem niedrigeren Risiko-Score von bis zu 0 (ergänzende Abb. 1 ). Das Elektrokardiogramm stimmte mit tachykardem Sinusrhythmus, rechtsatrialer Dilatation, RV-Hypertrophie und Belastung überein (ergänzende Abb. 2 ). Thoraxröntgen und Computertomographie (CT) zeigten eine starke Dilatation der RV und PAs, aber ein normales Lungenparenchym und keine Hinweise auf Thromben oder Venenverschlusserkrankungen (ergänzende Abb. 3 und 4 ). Pulmonale Angiogramme zeigten ein abnormes peripheres pulmonales Gefäßmuster, das durch eine sehr markante arterielle Schlängelung und Trübung des Kontrastmittels im peripheren Lungenkreislauf gekennzeichnet war; Letzteres kann durchgehend eine diffuse (prä)kapilläre Teleangiektasie und/oder sehr kleine arteriovenöse Fehlbildungen darstellen (ergänzende Abb. 5 und ergänzendes Video 1a,b ).Bei der Patientin wurde eine heterozygote Missense-Mutation im ACVRL1 -Gen festgestellt (c.1451 G>A, p.(Arg484Gln)). Diese Variante trat de novo auf und wurde zuvor bei sieben Patienten mit entweder isolierter PAH oder PAH plus HHT berichtet 11 , 12 , 13 , was sie dazu qualifiziert, vererbbare PAH und HHT Typ 2 zu haben.Nach der Diagnose (CATH #0) wurde der Patient mit einer dualen oralen Kombinationstherapie (Sildenafil und Bosentan) begonnen und zur Beurteilung der Lungentransplantation an die Medizinische Hochschule Hannover überwiesen 14 , 15 . Nach begrenztem Ansprechen auf die initiale duale orale Therapie wurde die gegen PAH gerichtete Pharmakotherapie modifiziert und umfasste orales Sildenafil, Macitentan, Spironolacton und inhalatives Iloprost. Da der systemische Blutdruck niedrig blieb (systolisch 80 mmHg), begannen wir nicht mit intravenösen Prostacyclin-Analoga. Unter diesen Maßnahmen wurde der Patient klinisch stabilisiert und die WHO-Funktionsklasse und 6MGT verbesserten sich (Ergänzungstabelle 1 ).Nichtsdestotrotz waren die echokardiographischen Variablen der Wechselwirkung zwischen rechtem Ventrikel und linkem Ventrikel (RV-LV) und der LV-Unterfüllung (RV/LV-endsystolisches Verhältnis, LV-endsystolischer Exzentrizitätsindex) und der Beschleunigungszeit der Pulmonalarterie (ein inverses Surrogat für den Schweregrad der PAH) unverändert stark unnormal. Die globale und longitudinale systolische RV-Funktion war reduziert. Aufgrund der düsteren Prognose wurde eine allogene HUCMSC-abgeleitete Therapie (Consented Compassionate Use) verfolgt.Dazu isolierten wir HUCMSCs 16 aus der Nabelschnur des jüngeren Geschwisters des Patienten und sammelten CM ( Methoden ). Der PAH-Patient erhielt eine Serie von fünf nicht GMP-zertifizierten, allogenen HUCMSC-CM-Infusionen über 6 Monate während zweier Krankenhausaufenthalte (Zeit 0 und Zeit 1) über einen intrapulmonalen Arterienkatheter und einen zentralen Venenkatheter. Alle HUCMSC-CM-Infusionen wurden sehr gut vertragen. Die Serum-CRP- und IL-6-Spiegel blieben normal, und der Patient erhielt keine antibiotischen, antiallergischen oder entzündungshemmenden Medikamente. Die Überwachung nach der Infusion erfolgte mindestens 24 Stunden, gefolgt von einer engmaschigen ambulanten Betreuung alle 1–6 Wochen. Der klinische Zustand und die invasive Hämodynamik wurden zu Studienbeginn (Zeitpunkt 0, CATH Nr. 1), nach 2 Monaten (Zeitpunkt 1, CATH Nr. 2) und nach 6 Monaten (Zeitpunkt 2, CATH Nr. 3) beurteilt (Abb. 1a ).Abb. 1: Die Behandlung mit HUCMSC-CM führt zu einer Verbesserung des Wachstums, der funktionellen Kapazität, der Risikobewertung und mehrerer hämodynamischer Variablen.
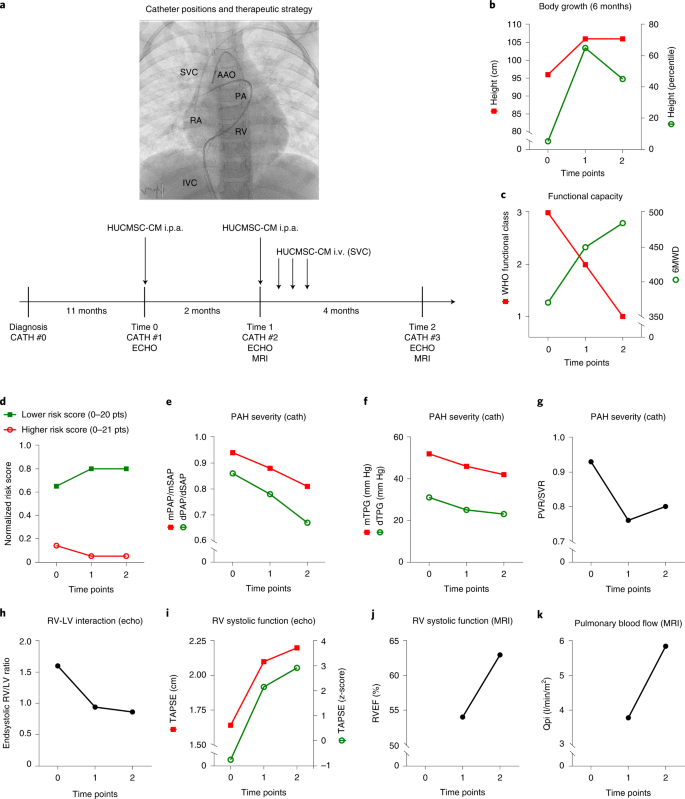 a , Vier Katheterisierungen (CATH Nr. 0, Nr. 1, Nr. 2 und Nr. 3) wurden im Abstand von 11 Monaten, 2 Monaten und 4 Monaten durchgeführt (in der Zeitachse als vertikale Balken dargestellt). Die PAH-Medikation wurde nicht innerhalb der 6 Monate vor CATH #1 (Zeitpunkt 0, an unserer Einrichtung) geändert, als HUCMSC-CM zum ersten Mal in den PAs verabreicht wurde. Auch die PAH-Medikation wurde danach nicht geändert. Während CATH Nr. 1 (Zeit 0) und CATH Nr. 2 (Zeit 1) wurden nach vollständiger invasiver hämodynamischer Beurteilung 200 ml HUCMSC-CM über 1 Stunde in die PAs infundiert (100 ml in die rechte PA über 30 Minuten; 100 ml in die linke PA über 30 Minuten). In der Woche nach CATH #2 (Zeitpunkt 1) wurden 200 ml HUCMSC-CM über einen zentralvenösen Zugang an den Tagen 1, 2 und 3 nach CATH #2 infundiert. Verbesserungen des Körperwachstums ( b ), der funktionellen Kapazität ( c), EPPVDN-Risikowerte ( d ) und morphologische und hämodynamische Schlüsseldaten ( e – k ), wie durch Herzkatheterisierung, Echokardiographie und kardiale Magnetresonanztomographie beurteilt, wurden zu den oben angegebenen Zeitpunkten (Zeitpunkt 0–2) generiert. Siehe
Methoden
für methodische Details. Katheterisierung, Rechts- und Linksherzkatheter; dTPG, diastolischer transpulmonaler Druckgradient; Echo, transthorakale Echokardiographie; MRI, kardiale Magnetresonanztomographie; mTPG, mittlerer transpulmonaler Druckgradient; Qpi, pulmonaler Blutflussindex; SVR, systemischer Gefäßwiderstand.
Quelldaten
Im Zeitraum zwischen Diagnose und Beginn der therapeutischen HUCMSC-CM-Intervention gab es im Wesentlichen keine Gewichtszunahme und kein Wachstum (Höhe) in 12 Monaten. Nach der ersten HUCMSC-CM-Infusion (Abb.
1a
) begann der Patient zu wachsen: +10 cm Länge in 3 Monaten (Gewinn vom 5. bis zum 65. Perzentil; Abb.
1b
). Darüber hinaus nahm die kardiopulmonale Belastungskapazität stark zu, wie anhand der WHO-Funktionsklasse (von 3 auf 1) und 6MWD (von 370 m auf 485 m; Abb.
1c
) beurteilt. Das Mädchen ist jetzt 6 Jahre alt und es geht ihr sehr gut, ohne Einschränkungen in der körperlichen Leistungsfähigkeit.In Übereinstimmung mit den klinischen Verbesserungen bestätigten die verbesserten EPPVDN-Risikowerte (Abb.
1d
), Herzkatheteruntersuchungen (Abb.
1e–g
) und Echokardiographiedaten (Abb.
1h,i
) die positive Wirkung der HUCMSC-CM-Behandlung: PA-Druck (PAP ) und PAP-Gradienten (Abb.
1e,f
) sowie das PVR/SVR-Verhältnis (Abb.
1g
) nahmen um 14–26 % ab (Abb.
1e–g
und Ergänzungstabelle
1
), was auf eine deutliche Abnahme des PAH-Schweregrads hinweist. Darüber hinaus zeigte die Echokardiographie, dass sich die RV-LV-Interaktion, beurteilt anhand des normalisierten endsystolischen RV/LV-Verhältnisses (Abb.
1h
) und der systolischen RV-Funktion (TAPSE; Abb.
1i
), stark verbessert hatte (ergänzende Abb.
7
und Zusatzvideo
2a,b
). Die Normalisierung der systolischen RV-Funktion und des pulmonalen Blutflusses wurden durch kardiale Magnetresonanztomographie zu Zeitpunkt 1 und Zeitpunkt 2 bestätigt (Abb.
1j, k
und Ergänzungstabelle
1
).Um die möglichen Mechanismen der von HUCMSC abgeleiteten Therapie zu untersuchen, führten wir eine Einzelzell-RNA-Sequenzierung (scRNA-seq) der subkultivierten MSCs durch, die verwendet wurden, um CM für die Compassionate-Use-Behandlung zu ernten (Abb.
2a
), Massenspektrometrie von MSC- CM (Abb.
2b
) und Proteinexpressionsassays des zu verschiedenen Zeitpunkten gesammelten Blutplasmas des Patienten (Abb.
2c–f
). scRNA-seq der subkultivierten HUCMSCs identifizierte vier funktionell unterschiedliche Zellsubpopulationen (Cluster). Die vier MSC-Subpopulationen sind in Abb.
2a
mit funktionellen Markierungen dargestellt, basierend auf der Pathway/Gene Ontology (GO)-Annotation ihrer Markergene (Cluster 0–3; Abb.
2a
, Ergänzungstabelle
2
und ergänzende Datentabelle
1
). Ergänzende Fig.
8
zeigt eine Expressions-Heatmap von drei Sätzen hochregulierter Gene, deren Expression die Zellen in die Subpopulationen aufteilt (oberste zehn pro Cluster gezeigt; keine hochregulierten Gene für Cluster 3). Insbesondere der Zellcluster 0 verstärkte das Transkriptom für Regeneration und Entzündungshemmung und sondert wahrscheinlich Moleküle ab, deren parakrine Wirkung positive Auswirkungen auf den rechten Herz-Lungen-Kreislauf hat.Abb. 2: scRNA-seq von HUCMSCs zeigt vier MSC-Cluster und ein Transkriptom, das für Regeneration, Entzündungshemmung, Zellzyklus und Stoffwechsel verbessert ist.
a , Vier Katheterisierungen (CATH Nr. 0, Nr. 1, Nr. 2 und Nr. 3) wurden im Abstand von 11 Monaten, 2 Monaten und 4 Monaten durchgeführt (in der Zeitachse als vertikale Balken dargestellt). Die PAH-Medikation wurde nicht innerhalb der 6 Monate vor CATH #1 (Zeitpunkt 0, an unserer Einrichtung) geändert, als HUCMSC-CM zum ersten Mal in den PAs verabreicht wurde. Auch die PAH-Medikation wurde danach nicht geändert. Während CATH Nr. 1 (Zeit 0) und CATH Nr. 2 (Zeit 1) wurden nach vollständiger invasiver hämodynamischer Beurteilung 200 ml HUCMSC-CM über 1 Stunde in die PAs infundiert (100 ml in die rechte PA über 30 Minuten; 100 ml in die linke PA über 30 Minuten). In der Woche nach CATH #2 (Zeitpunkt 1) wurden 200 ml HUCMSC-CM über einen zentralvenösen Zugang an den Tagen 1, 2 und 3 nach CATH #2 infundiert. Verbesserungen des Körperwachstums ( b ), der funktionellen Kapazität ( c), EPPVDN-Risikowerte ( d ) und morphologische und hämodynamische Schlüsseldaten ( e – k ), wie durch Herzkatheterisierung, Echokardiographie und kardiale Magnetresonanztomographie beurteilt, wurden zu den oben angegebenen Zeitpunkten (Zeitpunkt 0–2) generiert. Siehe
Methoden
für methodische Details. Katheterisierung, Rechts- und Linksherzkatheter; dTPG, diastolischer transpulmonaler Druckgradient; Echo, transthorakale Echokardiographie; MRI, kardiale Magnetresonanztomographie; mTPG, mittlerer transpulmonaler Druckgradient; Qpi, pulmonaler Blutflussindex; SVR, systemischer Gefäßwiderstand.
Quelldaten
Im Zeitraum zwischen Diagnose und Beginn der therapeutischen HUCMSC-CM-Intervention gab es im Wesentlichen keine Gewichtszunahme und kein Wachstum (Höhe) in 12 Monaten. Nach der ersten HUCMSC-CM-Infusion (Abb.
1a
) begann der Patient zu wachsen: +10 cm Länge in 3 Monaten (Gewinn vom 5. bis zum 65. Perzentil; Abb.
1b
). Darüber hinaus nahm die kardiopulmonale Belastungskapazität stark zu, wie anhand der WHO-Funktionsklasse (von 3 auf 1) und 6MWD (von 370 m auf 485 m; Abb.
1c
) beurteilt. Das Mädchen ist jetzt 6 Jahre alt und es geht ihr sehr gut, ohne Einschränkungen in der körperlichen Leistungsfähigkeit.In Übereinstimmung mit den klinischen Verbesserungen bestätigten die verbesserten EPPVDN-Risikowerte (Abb.
1d
), Herzkatheteruntersuchungen (Abb.
1e–g
) und Echokardiographiedaten (Abb.
1h,i
) die positive Wirkung der HUCMSC-CM-Behandlung: PA-Druck (PAP ) und PAP-Gradienten (Abb.
1e,f
) sowie das PVR/SVR-Verhältnis (Abb.
1g
) nahmen um 14–26 % ab (Abb.
1e–g
und Ergänzungstabelle
1
), was auf eine deutliche Abnahme des PAH-Schweregrads hinweist. Darüber hinaus zeigte die Echokardiographie, dass sich die RV-LV-Interaktion, beurteilt anhand des normalisierten endsystolischen RV/LV-Verhältnisses (Abb.
1h
) und der systolischen RV-Funktion (TAPSE; Abb.
1i
), stark verbessert hatte (ergänzende Abb.
7
und Zusatzvideo
2a,b
). Die Normalisierung der systolischen RV-Funktion und des pulmonalen Blutflusses wurden durch kardiale Magnetresonanztomographie zu Zeitpunkt 1 und Zeitpunkt 2 bestätigt (Abb.
1j, k
und Ergänzungstabelle
1
).Um die möglichen Mechanismen der von HUCMSC abgeleiteten Therapie zu untersuchen, führten wir eine Einzelzell-RNA-Sequenzierung (scRNA-seq) der subkultivierten MSCs durch, die verwendet wurden, um CM für die Compassionate-Use-Behandlung zu ernten (Abb.
2a
), Massenspektrometrie von MSC- CM (Abb.
2b
) und Proteinexpressionsassays des zu verschiedenen Zeitpunkten gesammelten Blutplasmas des Patienten (Abb.
2c–f
). scRNA-seq der subkultivierten HUCMSCs identifizierte vier funktionell unterschiedliche Zellsubpopulationen (Cluster). Die vier MSC-Subpopulationen sind in Abb.
2a
mit funktionellen Markierungen dargestellt, basierend auf der Pathway/Gene Ontology (GO)-Annotation ihrer Markergene (Cluster 0–3; Abb.
2a
, Ergänzungstabelle
2
und ergänzende Datentabelle
1
). Ergänzende Fig.
8
zeigt eine Expressions-Heatmap von drei Sätzen hochregulierter Gene, deren Expression die Zellen in die Subpopulationen aufteilt (oberste zehn pro Cluster gezeigt; keine hochregulierten Gene für Cluster 3). Insbesondere der Zellcluster 0 verstärkte das Transkriptom für Regeneration und Entzündungshemmung und sondert wahrscheinlich Moleküle ab, deren parakrine Wirkung positive Auswirkungen auf den rechten Herz-Lungen-Kreislauf hat.Abb. 2: scRNA-seq von HUCMSCs zeigt vier MSC-Cluster und ein Transkriptom, das für Regeneration, Entzündungshemmung, Zellzyklus und Stoffwechsel verbessert ist.
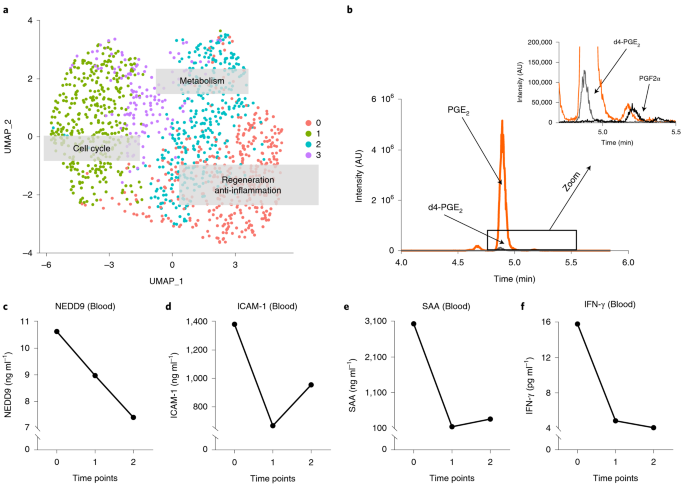 a , UMAP-Graph (Uniform Manifold Approximation and Projection) der ersten beiden UMAP-Dimensionen zeigt vier Cluster von MSCs. Basierend auf der Pathway/GO-Annotation ihrer Markergene wurden die funktionellen Markierungen dem Diagramm hinzugefügt. MSC-ähnliche Zellen wurden aus der menschlichen Nabelschnur (Wharton's Jelly) nach der Geburt eines voll ausgetragenen Säuglings (38. Schwangerschaftswoche) isoliert. Nach der MSC-Kultur wurden MSCs in Passage 3 geerntet, um sich einer scRNA-seq zu unterziehen, wie in den
Methoden
beschrieben . b , LC-MS-Chromatogramm mit den massenspektrometrischen Spuren von PGE 2 , PGF 2α und dem internen Standard d4-PGE 2 . c – f, EDTA-Plasmakonzentrationen von NEDD9, ICAM-1, SAA und IFN-γ (Mittelwert der Konzentrationen, die nahezu gleichzeitig in SVC, IVC und RA gemessen wurden). AU, willkürliche Einheiten.
Quelldaten
Basierend auf früheren präklinischen Stammzellstudien
17
,
18
,
19
stellten wir die Hypothese auf, dass die verstärkte Produktion von Prostaglandin E2 (PGE 2 ) eine wichtige regenerative und immunmodulatorische Komponente
20
bei HUCMSC-CM sein könnte. Tatsächlich zeigten die scRNA-seq-Daten, dass die Gene, die zwei PGE 2 -Syntheseenzyme ( PTGES2 und PTGES3 ) kodieren, sowie PTGS2 ( COX2 , ein Gen, das an der Umwandlung von Arachidonsäure (AA) in PGH 2 beteiligt ist, das für die Produktion von PGE 2 erforderlich ist ) wurden in den meisten HUCMSCs exprimiert, im Gegensatz zu den Enzymen, die PGI 2 synthetisierenund PGD 2 ( PTGIS und PTGDS schwach in nur wenigen Zellen exprimiert; ergänzende Fig.
9
). In Übereinstimmung mit unseren scRNA-seq-Daten detektierte die Analyse der HUCMSC-CM-Prostaglandine ein größeres PGE 2 -Signal, aber nur minimale Mengen an PGF 2α (
2b
) und kein PGD 2 , was insgesamt darauf hindeutet, dass HUCMSC-CM-sekretiertes PGE 2 sein könnte zur positiven Wirkung des HUCMSC-CM auf unseren PAH-Patienten beitragen.Die von HUCMSC abgeleitete Therapie senkte die Blutplasmamarker des Patienten für vaskuläre (endotheliale) Fibrose (NEDD9 (Ref.
21
)), vaskuläre Verletzung (ICAM-1) und Entzündung (SAA; IFN-γ) (Abb.
2c–f
). Diese Ergebnisse stimmen mit unseren scRNA-seq-Daten (PGE 2 - Synthesegene) und der nachfolgenden Validierung in kultivierten Zellen und CM (Einzelzell-Transkriptom, Proteom und Prostaglandine) aus mehreren Nabelschnüren überein, wie unten beschrieben (Abb.
3
).Abb. 3: Umfassende Analyse von kultivierten Zellen und CM (Einzelzell-Transkriptom, Proteom und Prostaglandine) zeigt potenzielle Mechanismen auf, die die regenerativen Wirkungen von HUCMSC-CM erklären.
a , UMAP-Graph (Uniform Manifold Approximation and Projection) der ersten beiden UMAP-Dimensionen zeigt vier Cluster von MSCs. Basierend auf der Pathway/GO-Annotation ihrer Markergene wurden die funktionellen Markierungen dem Diagramm hinzugefügt. MSC-ähnliche Zellen wurden aus der menschlichen Nabelschnur (Wharton's Jelly) nach der Geburt eines voll ausgetragenen Säuglings (38. Schwangerschaftswoche) isoliert. Nach der MSC-Kultur wurden MSCs in Passage 3 geerntet, um sich einer scRNA-seq zu unterziehen, wie in den
Methoden
beschrieben . b , LC-MS-Chromatogramm mit den massenspektrometrischen Spuren von PGE 2 , PGF 2α und dem internen Standard d4-PGE 2 . c – f, EDTA-Plasmakonzentrationen von NEDD9, ICAM-1, SAA und IFN-γ (Mittelwert der Konzentrationen, die nahezu gleichzeitig in SVC, IVC und RA gemessen wurden). AU, willkürliche Einheiten.
Quelldaten
Basierend auf früheren präklinischen Stammzellstudien
17
,
18
,
19
stellten wir die Hypothese auf, dass die verstärkte Produktion von Prostaglandin E2 (PGE 2 ) eine wichtige regenerative und immunmodulatorische Komponente
20
bei HUCMSC-CM sein könnte. Tatsächlich zeigten die scRNA-seq-Daten, dass die Gene, die zwei PGE 2 -Syntheseenzyme ( PTGES2 und PTGES3 ) kodieren, sowie PTGS2 ( COX2 , ein Gen, das an der Umwandlung von Arachidonsäure (AA) in PGH 2 beteiligt ist, das für die Produktion von PGE 2 erforderlich ist ) wurden in den meisten HUCMSCs exprimiert, im Gegensatz zu den Enzymen, die PGI 2 synthetisierenund PGD 2 ( PTGIS und PTGDS schwach in nur wenigen Zellen exprimiert; ergänzende Fig.
9
). In Übereinstimmung mit unseren scRNA-seq-Daten detektierte die Analyse der HUCMSC-CM-Prostaglandine ein größeres PGE 2 -Signal, aber nur minimale Mengen an PGF 2α (
2b
) und kein PGD 2 , was insgesamt darauf hindeutet, dass HUCMSC-CM-sekretiertes PGE 2 sein könnte zur positiven Wirkung des HUCMSC-CM auf unseren PAH-Patienten beitragen.Die von HUCMSC abgeleitete Therapie senkte die Blutplasmamarker des Patienten für vaskuläre (endotheliale) Fibrose (NEDD9 (Ref.
21
)), vaskuläre Verletzung (ICAM-1) und Entzündung (SAA; IFN-γ) (Abb.
2c–f
). Diese Ergebnisse stimmen mit unseren scRNA-seq-Daten (PGE 2 - Synthesegene) und der nachfolgenden Validierung in kultivierten Zellen und CM (Einzelzell-Transkriptom, Proteom und Prostaglandine) aus mehreren Nabelschnüren überein, wie unten beschrieben (Abb.
3
).Abb. 3: Umfassende Analyse von kultivierten Zellen und CM (Einzelzell-Transkriptom, Proteom und Prostaglandine) zeigt potenzielle Mechanismen auf, die die regenerativen Wirkungen von HUCMSC-CM erklären.
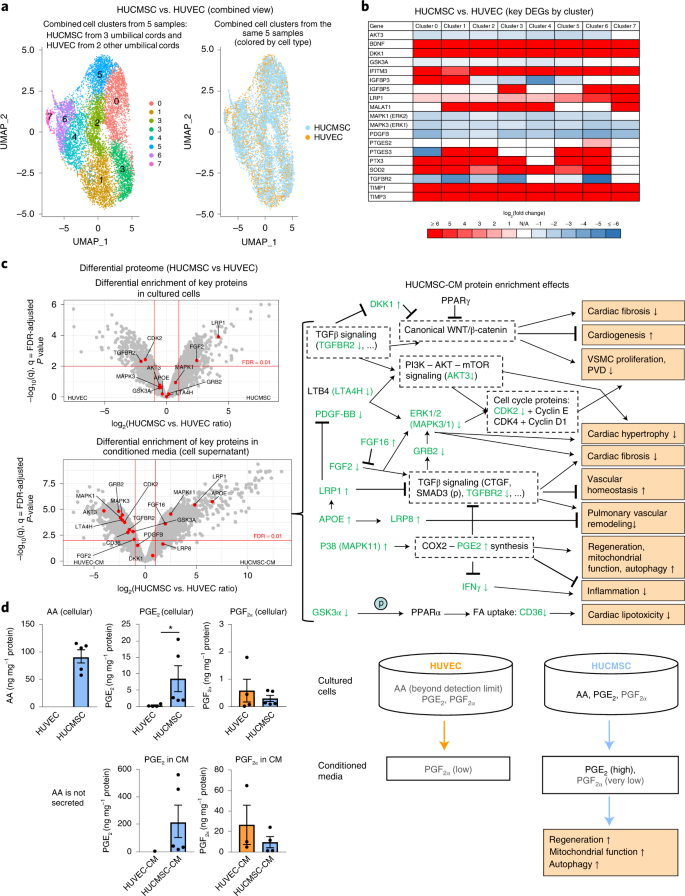 a . Einzelzell-RNA-Expressionsanalyse identifizierte acht Zellcluster mit unterschiedlichen Expressionsprofilen, die sowohl HUCMSC- als auch HUVEC-Zellen gemeinsam sind. Diese Cluster wurden für die differenzielle Genexpressionsanalyse (HUCMSC versus HUVEC) verwendet. Stichprobenumfänge: HUCMSC, n = 3 (Passage 6); HUVEC, n = 2 (Passage 6). Alle Schnüre wurden von nicht verwandten Spendern geerntet. b , Die Wärmekarte der Schlüsselgene , die möglicherweise zu regenerativen Wirkungen von HUCMSCs in allen acht in a gezeigten Clustern beitragen . Die Auswahl dieser Gene basierte auf (1) scRNA-seq-Analyse der HUCMSCs, deren CM bei der Behandlung des Falls verwendet wurde; (2) Ergebnisse der in c gezeigten Proteomikanalyse ; und (3) Assoziation mit der Synthese von PGE 2(das für seine Regenerationsfähigkeit bekannte Prostaglandin). c , Die Vulkandiagramme zeigen die Verteilung unterschiedlich angereicherter Proteine (HUCMSC, n = 5 versus HUVEC, n = 4). Die für das regenerative Potenzial relevanten Schlüsselproteine sind als rote Punkte dargestellt. Die Proteine, die über dem FDR-Schwellenwert (< 0,01) lagen und den Schwellenwert für die Effektstärke überschritten (0,5 > HUCMSC- versus HUVEC-Verhältnis > 2), wurden als signifikant angesehen. Die Konzentrationen von CM-Proteinen wichen von ihren Spiegeln in kultivierten Zellen ab (in den meisten Fällen erhöhte sich der Grad der differentiellen Anreicherung). Auf der rechten Seite zeigen wir mögliche Auswirkungen der Anreicherung der Schlüsselproteine im CM. Grün dargestellte Proteine wurden in unseren Daten beobachtet und entsprechen den im Vulkanplot markierten Proteinen (↑ = Hochregulierung, ↓ = Herunterregulierung, (p) = Phosphorylierung, fette Schrift zeigt FDR-bereinigtes P < 0,01 an). Das rechte Feld zeigt wahrscheinliche HUCMSC-CM-Proteinanreicherungseffekte. d, ergab die LC-MS-Prostaglandinanalyse, dass der Hauptunterschied zwischen HUCMSC- ( n = 5) und HUVEC- ( n = 4) Proben in den signifikant höheren HUCMSC-PGE 2 -Spiegeln (sowohl in Zellen als auch in CM) und höheren AA-Spiegeln in HUCMSCs bestand , während die HUVEC AA-Konzentrationen unter der Nachweisgrenze lagen. Da AA ein Vorläufer von PGE 2 ist, ist die Hochregulierung von PGE 2 in HUCMSCs ( P = 0,0159) wahrscheinlich mit dem höheren HUCMSC-AA-Spiegel verbunden. Bemerkenswerterweise trägt PGE 2 stark zu den parakrinen regenerativen und immunmodulatorischen Wirkungen von MSCs in präklinischen In-vivo- und In-vitro-Studien bei (z. B. PGE 2-induzierte Unterdrückung von IFN-γ- und TGFβ-Signalen). Spuren von PGF 2α wurden auch ohne signifikante Anreicherung in jeder der Gruppen nachgewiesen. Die Werte in Balkendiagrammwerten werden als Mittelwert ± SEM ausgedrückt; zweiseitiger Mann-Whitney- U - Test wurde verwendet (* P < 0,05 und ** P < 0,01). DEG, differentiell exprimiertes Gen.
Quelldaten
Wir haben unsere Einzelzell-RNA-Expressionsanalyse um HUCMSCs (drei Nabelschnüre) und HUVEC-Kontrollen (zwei Nabelschnüre) erweitert und acht Zellcluster mit unterschiedlichen Expressionsprofilen identifiziert (Abb.
3a
).Basierend auf der Analyse von unterschiedlich exprimierten Genen in diesen acht Clustern (HUCMSC versus HUVEC; ergänzende Datentabellen
2
–
9
) haben wir festgestellt, dass im Allgemeinen alle acht Cluster Expressionsprofile aufwiesen, die die vorteilhafte Rolle von HUCMSCs bestätigen – beispielsweise die Synthese von PGE2 und viele sezernierte Proteine (zum Beispiel DKK1, LRP1 und TGFBR2), die für ihre Rolle in regenerativen Wegen bekannt sind (
3b
). Die Konzentrationen von CM-Proteinen wichen von ihren intrazellulären Spiegeln in kultivierten Zellen in einer Weise ab, die in den meisten Fällen den Grad der differentiellen Anreicherung erhöhte (Abb.
3c
und ergänzende Datentabellen
10
und
11
).Die Analyse von Prostaglandinen, einschließlich des Vorläufers AA (
3d
), zeigte viel höhere Konzentrationen von HUCMSC PGE 2 (sowohl in Zellen als auch CM) und höhere Konzentrationen von AA in HUCMSCs im Vergleich zu HUVECs (die HUVEC AA-Konzentrationen lagen unter der Nachweisgrenze). Da AA ein Vorläufer von PGE 2 ist, ist die Hochregulierung von PGE 2 in HUCMSCs wahrscheinlich mit dem höheren HUCMSC-AA-Spiegel verbunden. Spuren von PGF 2α wurden auch ohne signifikante Anreicherung in jeder der Gruppen nachgewiesen. Zusammengenommen identifizierte die Flüssigkeitschromatographie-Massenspektrometrie (LC-MS)-Analyse sehr hohe Konzentrationen von PGE 2 in HUCMSCs und HUCMSC-CM, aber nicht in HUVECs oder HUVEC-CM (Abb.
3d
und ergänzende Datentabellen).
12
und
13
), höchstwahrscheinlich aufgrund einer verstärkten AA-PGE 2 -Synthese in HUCMSCs.DiskussionHier berichten wir unseres Wissens über die erste Anwendung von HUCMSC-CM am Menschen zur Behandlung schwerer, fortschreitender PAH bei einem Patienten. Serielle Infusionen von HUCMSC-CM führten nach 6 Monaten zu einer deutlichen klinischen und hämodynamischen Verbesserung und zeigten keine unerwünschten Ereignisse. Das HUCMSC-Transkriptom (von drei Nabelschnüren, nicht verwandte Spender) deutete auf eine Verbesserung der Regeneration, der mitochondrialen Funktion, der Autophagie und der entzündungshemmenden Wege hin. Die Proteomikanalyse ergab, dass die in HUCMSC-CM unterschiedlich angereicherten Proteine mehrere Schlüsselwege modulieren, um (1) Herzfibrose und -hypertrophie, Proliferation glatter Gefäßmuskelzellen (VSMC), Lungengefäßumbau, Entzündung und kardiale Lipotoxizität zu reduzieren und (2) die Kardiogenese zu erhöhen, vaskuläre Homöostase, Regeneration, Mitochondrienfunktion und Autophagie.2 -Signalisierung, die vom zellulären AA-Metabolismus in HUCMSCs abgeleitet ist.Bemerkenswerterweise senkte die von HUCMSC abgeleitete Therapie etablierte Blutplasmamarker für vaskuläre (endotheliale) Fibrose (NEDD9)
21
, vaskuläre Verletzung (ICAM-1) und Entzündung (Serum-Amyloid A (SAA) und IFN-γ) bei diesem Patienten. NEDD9 zielt auf Kollagen Typ 3 A1 ab und fördert die endotheliale Fibrose bei experimenteller PAH
21
. Darüber hinaus interagiert NEDD9 mit P-Selectin und fördert die nachteilige Thrombozyten-Endothel-Adhäsion im Lungenkreislauf
22
. Die endotheliale NEDD9- Expression wird durch Aldosteron (unabhängig von TGFβ - Signalen) reguliert
21
und in fibrotischen Arteriolen von Patienten mit PAH erhöht
21
. Es wurde gezeigt, dass der Blutplasma-NEDD9 bei erwachsener PAH um das 1,8-fache erhöht ist und positiv mit prognostischen Variablen (PVR) und negativ mit der RV-Funktion (rechtsventrikuläre Ejektionsfraktion (RVEF)), der körperlichen Leistungsfähigkeit (6MWD) und der Lungentransplantation korreliert -freies Überleben
23
. Bemerkenswerterweise verhinderte die Hemmung von NEDD9 experimentelle PAH
21
.Wir haben zuvor gezeigt, dass Aldosteron, das NEDD9 in Endothelzellen reguliert
21
, mit dem Schweregrad der PAH im Blutplasma von Erwachsenen zunimmt
24
. Hier verringerte die HUCMSC-CM-Behandlung, insbesondere die erste Dosis, den zirkulierenden Gefäßverletzungsmarker ICAM-1, der bei PAH
24
und den entzündungsfördernden Mediatoren SAA und IFN-γ erhöht ist.Humane MSCs gelten als immunologisch inert, ebenso wie zellfreie HUCMSC-CM-Infusionen, wie hier gezeigt. Wir haben zuvor eine überlegene Regenerationsfähigkeit von aus neonatalem Gewebe gewonnenen MSCs (Nabelschnur und Plazenta) im Vergleich zu MSCs nachgewiesen, die aus erwachsenen menschlichen Geweben (Knochenmark, peripheres Blut und Fettgewebe) isoliert wurden
20
. Eine langfristige Gewebetransplantation von MSCs wurde jedoch nie in vorklinischen oder klinischen Studien nachgewiesen. Dementsprechend wird vorgeschlagen, dass die wichtigsten vorteilhaften Wirkungen von MSCs parakriner Natur sind (Sekretion). MSC-abgeleitete extrazelluläre Vesikel (EVs)
25
, isoliert aus CM, haben eine ausgeprägte Effizienz bei Hyperoxie-induzierter neugeborener bronchopulmonaler Dysplasie bei Mäusen
26
und bei VEGFR2-blockierte/Hypoxie-exponierte Ratten mit PAH- und RV-Versagen
27
. In der letztgenannten Studie war die wiederholte Verabreichung von MSC-EVs aus Knochenmark innerhalb von Tagen am effizientesten bei der Umkehrung von PAH
27
. Es ist wahrscheinlich, dass Elektrofahrzeuge zu den positiven Wirkungen unserer HUCMSC-CM-Infusionen beigetragen haben. Allerdings sind die präklinisch verwendeten EVs häufig schlecht definiert
28
, was Vergleiche zwischen Studien und GMP-zertifizierte Therapien für den klinischen Einsatz zu einer schwierigen Aufgabe macht.Wir validierten unsere Fallergebnisse in einer anschließenden Multiple-Cord-Omics-Analyse von HUCMSCs im Vergleich zu HUVECs und ihrem Sekretom (CM): Auf molekularer Ebene bestätigten wir die Hochregulierung und sagten die Sekretion der wichtigsten Regenerations-/Proliferationsmoleküle voraus: IGFBP3 , IGFBP5 , BDNF , TIMP1 und TIMP3 (hochreguliert im regenerativen Zellcluster – d. h. Cluster 0 – der scRNA-seq-Ergebnisse des behandelten Patienten), die auch in den meisten Clustern der integrierten scRNA-seq-Analyse als hochreguliert gefunden wurden (HUCMSCs versus HUVECs; Abb
3b
) und positiv angereichert an HUCMSC-CM-Proteinen . Darüber hinaus sind die Ergebnisse der in Fig.
3c dargestellten Proteomics-Analyse dargestellt
schlagen eine Vielzahl von Mechanismen vor, die wahrscheinlich zu Verbesserungen der kardiovaskulären Funktion bei dem Patienten beitragen. Kurz gesagt, unsere Multi-Cord-Datenanalyse legt nahe, dass (1) die Herunterregulierung des TGFβ -Signalwegs die kanonische WNT-Signalgebung unterdrückt und dadurch die Herzfibrose reduziert
29
, die Kardiogenese fördert und die VSMC-Proliferation hemmt
30
; (2) Herunterregulierung von GSK3α verringert die Aufnahme von Fettsäuren (FA) (manifestiert durch Herunterregulierung von CD36) und verringert die Lipotoxizität
31
; (3) Unterdrückung der PI3K-AKT-mTOR- und ERK1/2-Signalgebung
32
reguliert VSMC-Proliferation und Herzhypertrophie herunter; (4) Hochregulierung von P38 (MAPK11) fördert regenerative Wirkungen durch Induktion der COX2-PGE 2 -Synthese
17
,
33
(auch unterstützt durch LC-MS-Analyse von Prostaglandinen, die hohe PGE 2 -Spiegel in HUCMSC, aber nicht in HUVEC sowohl in Zellen als auch in CM zeigen (Abb.
3d
und ergänzende
Datentabellen 12
und
13
)); (5) die Hochregulierung von APOE
34
, LRP1 (Ref.
35
) und FGF16 (Ref.
36
) und die Herunterregulierung von TGFBR2 und FGF2 (Ref.
36
) unterdrückt die TGFβ-Signalgebung, wodurch die
Herzfibrose reduziert wird 37
, wodurch eine vaskuläre Homöostase bei PAH hergestellt wird
8
; (6) Unterdrückung der ERK1/2-Signalisierung
32
hemmt die VSMC-Proliferation; und (7) die Hochregulierung der APOE-LRP8-Kaskade unterdrückt den pulmonalen Gefäßumbau
38
.Unsere erweiterte Multicord-LC-MS-Analyse von Prostaglandinen und AA bestätigte eine signifikante Hochregulation von PGE 2 (Zellen und CM), die ursprünglich im behandelten Fall identifiziert wurde (Abb.
3d
). Die Hochregulierung von PGE 2 wurde auch durch die integrierten scRNA-seq-Daten (Hochregulierung von PTGES2 und PTGES3 in HUCMSCs) und Zellproteomikdaten gestützt, wobei PTGES in allen HUVEC-Proben unter der Nachweisgrenze lag, aber in allen HUCMSC-Proben vorhanden war.Unsere Multiple-Cord-Omics-Ergebnisse untermauern die Gültigkeit der Komponentenbefunde und bestätigen, dass die Art und Weise, wie wir MSCs und CM herstellen, zwischen den verschiedenen Chargen konsistent ist – ein wichtiger Punkt im Hinblick auf Standardisierung und Reproduzierbarkeit.Die PGE 2 -Signalgebung stimuliert Stammzellen, beschädigtes Gewebe zu regenerieren
17
, erhöht die mitochondriale Funktion und Autophagie und verringert die IFN-γ- und TGFβ - Signalwege
19
, die bei PAH verstärkt werden
1
,
7
,
8
,
9
. Bemerkenswert ist, dass diese Ergebnisse vor kurzem in die Entwicklung von niedermolekularen 15-Prostagalandin-Dehydrogenase (15-PGDH)-Inhibitoren (SW033291) eingeflossen sind, die den Abbau von PGE 2 blockieren
39
; Klinische Studien stehen jedoch noch aus.Basierend sowohl auf den sehr hohen PGE 2 -Spiegeln, die wir in HUCMSC-CM gefunden haben, als auch auf der Induktion von drei PGE 2 -Syntheseenzymen in HUCMSCs schlagen wir vor, dass PGE 2 eine wichtige vorteilhafte Komponente von HUCMSC-CM mit vasodilatatorischen und regenerativen Eigenschaften ist PAH.Wir identifizierten bei unserem Patienten eine De-novo-Mißense-Mutation im ACVRL1 -Gen. Interessanterweise wurde diese spezielle ACVRL1 -Funktionsverlust-Mutation (c.1451 G>A, p.(Arg484Gln)) bei keinem Patienten mit HHT in Abwesenheit von PAH gefunden, was die Wirkung dieser Einzelnukleotid-Variante auf unterstreicht Lungengefäßentwicklung und Homöostase.Unsere Hauptabsicht war es, über die beeindruckenden Verbesserungen bei dem behandelten Kind und die wahrscheinlichen Mechanismen zu berichten, die durch unsere Multi-Omics-Analyse des Falls und die verfügbaren Nabelschnurproben aufgedeckt wurden. Hier demonstrieren wir die Sicherheit und Wirksamkeit einer von MSC abgeleiteten Therapie, jedoch nur bei einem Patienten. Zusätzliche Einschränkungen unserer Studie sind eine relativ kleine Anzahl von Validierungsfällen (drei Nabelschnüre). Weitere Studien sind für eine strenge Validierung unserer Ergebnisse erforderlich.Abschließend lässt sich sagen, dass serielle Infusionen von HUCMSC-CM bei einem jungen Patienten mit schwerer PAH zu einer deutlichen klinischen und hämodynamischen Verbesserung führten. Wir gehen davon aus, dass die von HUCMSC abgeleitete Therapie das Potenzial hat, eine wirksame Behandlung für die schwersten Formen der klinischen PAH zu werden. Weitere prospektive klinische Studien sind gerechtfertigt, um die Vorteile einer von HUCMSC abgeleiteten PAH-Therapie zu untersuchen.MethodenIsolierung von HUCMSC und HUVEC und Herstellung von Zell-abgeleitetem CMIsolierung, Kultur und Charakterisierung von primärem HUCMSC und HUVECMSC-ähnliche Zellen wurden aus der menschlichen Nabelschnur (Wharton's Jelly) nach der Geburt eines voll ausgetragenen (38. Schwangerschaftswoche) Säuglings (hier: jüngere Schwester, per Kaiserschnitt; Abb.
1
und
2
) und aus zusätzlicher menschlicher Nabelschnur isoliert Schnüre (Abb.
3
). Die Zellen wurden durch Explantatkultur in MSC-Wachstumsmedium
40
kultiviert . Kurz gesagt wurde Nabelschnurgewebe mehrere Male mit PBS gewaschen, um Blutzellen zu entfernen, in etwa 1,5 cm 3 große Stücke geschnitten und in MSC-Wachstumsmedium (&agr;MEM, Invitrogen), ergänzt mit 15 % allogenem humanem AB-Serum (HS ), 100 U ml –1 Penicillin, 100 mg ml –1von Streptomycin und 2 mM L-Glutamin bei 37°C in einer befeuchteten Atmosphäre mit 5% CO 2 . Die Explantatkultur wurde 15 Tage lang durchgeführt. Der Auswuchs einer adhärenten angereicherten MSC-Population wurde durch Behandlung mit Accutase (Capricorn Scientific) gemäß dem Protokoll des Herstellers für 5 Minuten bei 37 °C geerntet. Die Zellen wurden bei 320 g für 5 Minuten zentrifugiert, in MSC-Kulturmedium (&agr;MEM), ergänzt mit 10 % HS, 100 U ml –1 Penicillin, 100 mg ml –1 Streptomycin und 2 mM L-Glutamin, bei 37° resuspendiert C in einer befeuchteten Atmosphäre mit 5 % CO 2 und bei einer Dichte von 4.000 Zellen pro cm 2 kultiviert. Das Ernten und die Subkultivierung in entsprechenden Passagen erfolgte nach Behandlung mit Accutase (Capricorn Scientific) bei 37°C für 3 Minuten.Kontinuierlich proliferierende MSCs wurden geerntet und auf Zellzyklusprogression und Zelloberflächenmarkerexpression durch Durchflusszytometrie
40
analysiert . Neben nachweisbaren G1-, S- und G2/M-Phasen wurde das Vorhandensein von CD73, CD90 und CD105 bei gleichzeitiger Abwesenheit von CD14, CD31, CD34, CD45 und HLA-DR durch FACS-Analyse gemäß dem Vorschlag der International Society for Cellular getestet Therapie als eines der Minimalkriterien für die MSC-Charakterisierung
41
. HUVECs wurden von PromoCell (Kat.-Nr. C-12200, Heidelberg, Deutschland) bezogen und gemäß den Anweisungen des Herstellers (PromoCell Instruction Manual) bis P3 oder P6 (scRNA-seq) subkultiviert. Für Proteom- und LC-MS-Analysen wurde CM von HUCMSCs und HUVECs nach 3 Waschschritten in geeignetem serumfreiem Medium und Inkubation der Kulturen in entsprechendem serumfreiem Medium für 36 h gesammelt, gefolgt von Zentrifugation (3.185 g für 10 Minuten). .Herstellung von Zell-abgeleitetem CM aus HUCMSC zur intravaskulären InfusionNach der MSC-Kultur in den Passagen 2 und 3 in der subkonfluenten Wachstumsphase wurde der serumfreie Überstand als CM nach 36 Stunden geerntet, zentrifugiert (3,185 gfür 10 Minuten), negativ auf Bakterien- und Mykoplasmenkontamination getestet und bei −80 °C kryokonserviert. In der Nacht vor der Injektion (etwa 12–14 Stunden) wurde das HUCMSC-CM vorsichtig bei 4 °C aufgetaut und dann auf Raumtemperatur vorgewärmt. Für die HUCMSC-CM-Infusionen wurden bestimmte Filter verwendet: Zeit 0, CATH #1 (ipa): Dosis 1, 200 ml CM, Sterifix 0,2-μm-Filter (B. Braun, 4099303); Zeitpunkt 1, CATH #2 Dosis 2, ipa: 200 ml CM, Transfusionsfilter 200 µm (B. Braun, 8270066SP). Zeit 1, Dosis 3, SVC; 200 ml KM, Transfusionsfilter 200 µm (B. Braun, 8270066SP). Zeitpunkt 1, Dosis 4, SVC: 200 ml KM, Minisart 0,2 µm Filter (Sartorius/Th. Geyer, 90491011), Transfusionsfilter 200 µm (B. Braun). Zeitpunkt 1, Dosis 5, SVC: 200 ml KM, Minisart 0,2 µm Filter (Sartorius/Th. Geyer, 90491011), Transfusionsfilter 200 µm (B. Braun).Herzkatheterisierungen und Infusionen von HUCMSC-CMNach der Diagnose (CATH #0) wurden alle nachfolgenden Herzkatheteruntersuchungen an der Medizinischen Hochschule Hannover im Dezember 2019 (Zeitpunkt 0, CATH #1), Februar 2020 (Zeitpunkt 1, Cath #2) und Mai 2020 (Zeitpunkt 2, CATH #3) durchgeführt ) (Abb.
1a
und Ergänzungstabelle
1
). Die PAH-gerichtete Medikation wurde in den 6 Monaten vor CATH #1, als zum ersten Mal HUCMSC-CM in den PAs verabreicht wurde, nicht geändert und danach nicht geändert.Während CATH Nr. 1 und CATH Nr. 2 wurden nach vollständiger invasiver hämodynamischer Beurteilung 200 ml HUCMSC-CM in die PAs (ipa) über 1 Stunde infundiert – d. h. 100 ml in die rechte Pulmonalarterie (RPA) über 30 Minuten und 100 ml in die linke Pulmonalarterie (LPA) über 30 Minuten. In der Woche nach CATH Nr. 2 wurden 200 ml HUCMSC-CM über einen zentralvenösen Zugang über 60 Minuten an den Tagen 1, 2 und 3 nach CATH Nr. 2 infundiert (Abb.
1a
und ergänzende Abb.
6
).Mikrobiologische und immunologische Tests von HUCMSC, HUCMSC-CM, Empfänger (Patient), Spender (Schwester) und MutterHUCMSC/HUCMSC-CM (Zellkulturüberstand)Kultur 10–14 Tage negativ für Bakterienwachstum (aerob und anaerob)SpenderHLA-Typen:DNA-Typen HLA-1: A*30, A*68, B*13, B*18, C*06, C*12DNA-Typen HLA-2: DRB3*pos., DQB1*03HBs-Antigen-negativ, Anti-HBC-negativ, Anti-HBS 28 IE l –1Anti-HCV-negativ, CMV-IgM-negativ, CMV-IgG 606 U ml −1EBV-IgG 571 E ml –1 , VCA IgM negativ, EBNA1-IgG 141 E ml –1Toxoplasma-Suchtest negativTreponema pallidum IA-Test negativHIV-AK1/2, p24-Ag-negativEmpfängerBlutgruppe 0 Rh positiv, CcDD.Ee, K-, unregelmäßige Erythrozyten-Antikörper negativHLA-Typen:DNA-Typen HLA-1: A*24, A*68, B*13, B*18, C*06, C*12DNA-Typen HLA-2: DRB1*07, DRB1*11, DRB3*pos., DRB4*pos., DQB1*02, DQB1*03GvH-Konstellation (MSC-Transplantat versus Patient): A*24, C*04, DRB1*07, DQB1*02HvG-Konstellation (Patient gegen MSC-Transplantation): A*68, C*06Mycoplasma-IgM-negativ, Mycoplasma-IgG-negativDiagnosekriterien von Curaçao für HHT (Osler-Weber-Rendu-Syndrom) gemäß internationalen Richtlinien (2000 und 2020)
42
,
43
Zu diesen Kriterien gehören
a . Einzelzell-RNA-Expressionsanalyse identifizierte acht Zellcluster mit unterschiedlichen Expressionsprofilen, die sowohl HUCMSC- als auch HUVEC-Zellen gemeinsam sind. Diese Cluster wurden für die differenzielle Genexpressionsanalyse (HUCMSC versus HUVEC) verwendet. Stichprobenumfänge: HUCMSC, n = 3 (Passage 6); HUVEC, n = 2 (Passage 6). Alle Schnüre wurden von nicht verwandten Spendern geerntet. b , Die Wärmekarte der Schlüsselgene , die möglicherweise zu regenerativen Wirkungen von HUCMSCs in allen acht in a gezeigten Clustern beitragen . Die Auswahl dieser Gene basierte auf (1) scRNA-seq-Analyse der HUCMSCs, deren CM bei der Behandlung des Falls verwendet wurde; (2) Ergebnisse der in c gezeigten Proteomikanalyse ; und (3) Assoziation mit der Synthese von PGE 2(das für seine Regenerationsfähigkeit bekannte Prostaglandin). c , Die Vulkandiagramme zeigen die Verteilung unterschiedlich angereicherter Proteine (HUCMSC, n = 5 versus HUVEC, n = 4). Die für das regenerative Potenzial relevanten Schlüsselproteine sind als rote Punkte dargestellt. Die Proteine, die über dem FDR-Schwellenwert (< 0,01) lagen und den Schwellenwert für die Effektstärke überschritten (0,5 > HUCMSC- versus HUVEC-Verhältnis > 2), wurden als signifikant angesehen. Die Konzentrationen von CM-Proteinen wichen von ihren Spiegeln in kultivierten Zellen ab (in den meisten Fällen erhöhte sich der Grad der differentiellen Anreicherung). Auf der rechten Seite zeigen wir mögliche Auswirkungen der Anreicherung der Schlüsselproteine im CM. Grün dargestellte Proteine wurden in unseren Daten beobachtet und entsprechen den im Vulkanplot markierten Proteinen (↑ = Hochregulierung, ↓ = Herunterregulierung, (p) = Phosphorylierung, fette Schrift zeigt FDR-bereinigtes P < 0,01 an). Das rechte Feld zeigt wahrscheinliche HUCMSC-CM-Proteinanreicherungseffekte. d, ergab die LC-MS-Prostaglandinanalyse, dass der Hauptunterschied zwischen HUCMSC- ( n = 5) und HUVEC- ( n = 4) Proben in den signifikant höheren HUCMSC-PGE 2 -Spiegeln (sowohl in Zellen als auch in CM) und höheren AA-Spiegeln in HUCMSCs bestand , während die HUVEC AA-Konzentrationen unter der Nachweisgrenze lagen. Da AA ein Vorläufer von PGE 2 ist, ist die Hochregulierung von PGE 2 in HUCMSCs ( P = 0,0159) wahrscheinlich mit dem höheren HUCMSC-AA-Spiegel verbunden. Bemerkenswerterweise trägt PGE 2 stark zu den parakrinen regenerativen und immunmodulatorischen Wirkungen von MSCs in präklinischen In-vivo- und In-vitro-Studien bei (z. B. PGE 2-induzierte Unterdrückung von IFN-γ- und TGFβ-Signalen). Spuren von PGF 2α wurden auch ohne signifikante Anreicherung in jeder der Gruppen nachgewiesen. Die Werte in Balkendiagrammwerten werden als Mittelwert ± SEM ausgedrückt; zweiseitiger Mann-Whitney- U - Test wurde verwendet (* P < 0,05 und ** P < 0,01). DEG, differentiell exprimiertes Gen.
Quelldaten
Wir haben unsere Einzelzell-RNA-Expressionsanalyse um HUCMSCs (drei Nabelschnüre) und HUVEC-Kontrollen (zwei Nabelschnüre) erweitert und acht Zellcluster mit unterschiedlichen Expressionsprofilen identifiziert (Abb.
3a
).Basierend auf der Analyse von unterschiedlich exprimierten Genen in diesen acht Clustern (HUCMSC versus HUVEC; ergänzende Datentabellen
2
–
9
) haben wir festgestellt, dass im Allgemeinen alle acht Cluster Expressionsprofile aufwiesen, die die vorteilhafte Rolle von HUCMSCs bestätigen – beispielsweise die Synthese von PGE2 und viele sezernierte Proteine (zum Beispiel DKK1, LRP1 und TGFBR2), die für ihre Rolle in regenerativen Wegen bekannt sind (
3b
). Die Konzentrationen von CM-Proteinen wichen von ihren intrazellulären Spiegeln in kultivierten Zellen in einer Weise ab, die in den meisten Fällen den Grad der differentiellen Anreicherung erhöhte (Abb.
3c
und ergänzende Datentabellen
10
und
11
).Die Analyse von Prostaglandinen, einschließlich des Vorläufers AA (
3d
), zeigte viel höhere Konzentrationen von HUCMSC PGE 2 (sowohl in Zellen als auch CM) und höhere Konzentrationen von AA in HUCMSCs im Vergleich zu HUVECs (die HUVEC AA-Konzentrationen lagen unter der Nachweisgrenze). Da AA ein Vorläufer von PGE 2 ist, ist die Hochregulierung von PGE 2 in HUCMSCs wahrscheinlich mit dem höheren HUCMSC-AA-Spiegel verbunden. Spuren von PGF 2α wurden auch ohne signifikante Anreicherung in jeder der Gruppen nachgewiesen. Zusammengenommen identifizierte die Flüssigkeitschromatographie-Massenspektrometrie (LC-MS)-Analyse sehr hohe Konzentrationen von PGE 2 in HUCMSCs und HUCMSC-CM, aber nicht in HUVECs oder HUVEC-CM (Abb.
3d
und ergänzende Datentabellen).
12
und
13
), höchstwahrscheinlich aufgrund einer verstärkten AA-PGE 2 -Synthese in HUCMSCs.DiskussionHier berichten wir unseres Wissens über die erste Anwendung von HUCMSC-CM am Menschen zur Behandlung schwerer, fortschreitender PAH bei einem Patienten. Serielle Infusionen von HUCMSC-CM führten nach 6 Monaten zu einer deutlichen klinischen und hämodynamischen Verbesserung und zeigten keine unerwünschten Ereignisse. Das HUCMSC-Transkriptom (von drei Nabelschnüren, nicht verwandte Spender) deutete auf eine Verbesserung der Regeneration, der mitochondrialen Funktion, der Autophagie und der entzündungshemmenden Wege hin. Die Proteomikanalyse ergab, dass die in HUCMSC-CM unterschiedlich angereicherten Proteine mehrere Schlüsselwege modulieren, um (1) Herzfibrose und -hypertrophie, Proliferation glatter Gefäßmuskelzellen (VSMC), Lungengefäßumbau, Entzündung und kardiale Lipotoxizität zu reduzieren und (2) die Kardiogenese zu erhöhen, vaskuläre Homöostase, Regeneration, Mitochondrienfunktion und Autophagie.2 -Signalisierung, die vom zellulären AA-Metabolismus in HUCMSCs abgeleitet ist.Bemerkenswerterweise senkte die von HUCMSC abgeleitete Therapie etablierte Blutplasmamarker für vaskuläre (endotheliale) Fibrose (NEDD9)
21
, vaskuläre Verletzung (ICAM-1) und Entzündung (Serum-Amyloid A (SAA) und IFN-γ) bei diesem Patienten. NEDD9 zielt auf Kollagen Typ 3 A1 ab und fördert die endotheliale Fibrose bei experimenteller PAH
21
. Darüber hinaus interagiert NEDD9 mit P-Selectin und fördert die nachteilige Thrombozyten-Endothel-Adhäsion im Lungenkreislauf
22
. Die endotheliale NEDD9- Expression wird durch Aldosteron (unabhängig von TGFβ - Signalen) reguliert
21
und in fibrotischen Arteriolen von Patienten mit PAH erhöht
21
. Es wurde gezeigt, dass der Blutplasma-NEDD9 bei erwachsener PAH um das 1,8-fache erhöht ist und positiv mit prognostischen Variablen (PVR) und negativ mit der RV-Funktion (rechtsventrikuläre Ejektionsfraktion (RVEF)), der körperlichen Leistungsfähigkeit (6MWD) und der Lungentransplantation korreliert -freies Überleben
23
. Bemerkenswerterweise verhinderte die Hemmung von NEDD9 experimentelle PAH
21
.Wir haben zuvor gezeigt, dass Aldosteron, das NEDD9 in Endothelzellen reguliert
21
, mit dem Schweregrad der PAH im Blutplasma von Erwachsenen zunimmt
24
. Hier verringerte die HUCMSC-CM-Behandlung, insbesondere die erste Dosis, den zirkulierenden Gefäßverletzungsmarker ICAM-1, der bei PAH
24
und den entzündungsfördernden Mediatoren SAA und IFN-γ erhöht ist.Humane MSCs gelten als immunologisch inert, ebenso wie zellfreie HUCMSC-CM-Infusionen, wie hier gezeigt. Wir haben zuvor eine überlegene Regenerationsfähigkeit von aus neonatalem Gewebe gewonnenen MSCs (Nabelschnur und Plazenta) im Vergleich zu MSCs nachgewiesen, die aus erwachsenen menschlichen Geweben (Knochenmark, peripheres Blut und Fettgewebe) isoliert wurden
20
. Eine langfristige Gewebetransplantation von MSCs wurde jedoch nie in vorklinischen oder klinischen Studien nachgewiesen. Dementsprechend wird vorgeschlagen, dass die wichtigsten vorteilhaften Wirkungen von MSCs parakriner Natur sind (Sekretion). MSC-abgeleitete extrazelluläre Vesikel (EVs)
25
, isoliert aus CM, haben eine ausgeprägte Effizienz bei Hyperoxie-induzierter neugeborener bronchopulmonaler Dysplasie bei Mäusen
26
und bei VEGFR2-blockierte/Hypoxie-exponierte Ratten mit PAH- und RV-Versagen
27
. In der letztgenannten Studie war die wiederholte Verabreichung von MSC-EVs aus Knochenmark innerhalb von Tagen am effizientesten bei der Umkehrung von PAH
27
. Es ist wahrscheinlich, dass Elektrofahrzeuge zu den positiven Wirkungen unserer HUCMSC-CM-Infusionen beigetragen haben. Allerdings sind die präklinisch verwendeten EVs häufig schlecht definiert
28
, was Vergleiche zwischen Studien und GMP-zertifizierte Therapien für den klinischen Einsatz zu einer schwierigen Aufgabe macht.Wir validierten unsere Fallergebnisse in einer anschließenden Multiple-Cord-Omics-Analyse von HUCMSCs im Vergleich zu HUVECs und ihrem Sekretom (CM): Auf molekularer Ebene bestätigten wir die Hochregulierung und sagten die Sekretion der wichtigsten Regenerations-/Proliferationsmoleküle voraus: IGFBP3 , IGFBP5 , BDNF , TIMP1 und TIMP3 (hochreguliert im regenerativen Zellcluster – d. h. Cluster 0 – der scRNA-seq-Ergebnisse des behandelten Patienten), die auch in den meisten Clustern der integrierten scRNA-seq-Analyse als hochreguliert gefunden wurden (HUCMSCs versus HUVECs; Abb
3b
) und positiv angereichert an HUCMSC-CM-Proteinen . Darüber hinaus sind die Ergebnisse der in Fig.
3c dargestellten Proteomics-Analyse dargestellt
schlagen eine Vielzahl von Mechanismen vor, die wahrscheinlich zu Verbesserungen der kardiovaskulären Funktion bei dem Patienten beitragen. Kurz gesagt, unsere Multi-Cord-Datenanalyse legt nahe, dass (1) die Herunterregulierung des TGFβ -Signalwegs die kanonische WNT-Signalgebung unterdrückt und dadurch die Herzfibrose reduziert
29
, die Kardiogenese fördert und die VSMC-Proliferation hemmt
30
; (2) Herunterregulierung von GSK3α verringert die Aufnahme von Fettsäuren (FA) (manifestiert durch Herunterregulierung von CD36) und verringert die Lipotoxizität
31
; (3) Unterdrückung der PI3K-AKT-mTOR- und ERK1/2-Signalgebung
32
reguliert VSMC-Proliferation und Herzhypertrophie herunter; (4) Hochregulierung von P38 (MAPK11) fördert regenerative Wirkungen durch Induktion der COX2-PGE 2 -Synthese
17
,
33
(auch unterstützt durch LC-MS-Analyse von Prostaglandinen, die hohe PGE 2 -Spiegel in HUCMSC, aber nicht in HUVEC sowohl in Zellen als auch in CM zeigen (Abb.
3d
und ergänzende
Datentabellen 12
und
13
)); (5) die Hochregulierung von APOE
34
, LRP1 (Ref.
35
) und FGF16 (Ref.
36
) und die Herunterregulierung von TGFBR2 und FGF2 (Ref.
36
) unterdrückt die TGFβ-Signalgebung, wodurch die
Herzfibrose reduziert wird 37
, wodurch eine vaskuläre Homöostase bei PAH hergestellt wird
8
; (6) Unterdrückung der ERK1/2-Signalisierung
32
hemmt die VSMC-Proliferation; und (7) die Hochregulierung der APOE-LRP8-Kaskade unterdrückt den pulmonalen Gefäßumbau
38
.Unsere erweiterte Multicord-LC-MS-Analyse von Prostaglandinen und AA bestätigte eine signifikante Hochregulation von PGE 2 (Zellen und CM), die ursprünglich im behandelten Fall identifiziert wurde (Abb.
3d
). Die Hochregulierung von PGE 2 wurde auch durch die integrierten scRNA-seq-Daten (Hochregulierung von PTGES2 und PTGES3 in HUCMSCs) und Zellproteomikdaten gestützt, wobei PTGES in allen HUVEC-Proben unter der Nachweisgrenze lag, aber in allen HUCMSC-Proben vorhanden war.Unsere Multiple-Cord-Omics-Ergebnisse untermauern die Gültigkeit der Komponentenbefunde und bestätigen, dass die Art und Weise, wie wir MSCs und CM herstellen, zwischen den verschiedenen Chargen konsistent ist – ein wichtiger Punkt im Hinblick auf Standardisierung und Reproduzierbarkeit.Die PGE 2 -Signalgebung stimuliert Stammzellen, beschädigtes Gewebe zu regenerieren
17
, erhöht die mitochondriale Funktion und Autophagie und verringert die IFN-γ- und TGFβ - Signalwege
19
, die bei PAH verstärkt werden
1
,
7
,
8
,
9
. Bemerkenswert ist, dass diese Ergebnisse vor kurzem in die Entwicklung von niedermolekularen 15-Prostagalandin-Dehydrogenase (15-PGDH)-Inhibitoren (SW033291) eingeflossen sind, die den Abbau von PGE 2 blockieren
39
; Klinische Studien stehen jedoch noch aus.Basierend sowohl auf den sehr hohen PGE 2 -Spiegeln, die wir in HUCMSC-CM gefunden haben, als auch auf der Induktion von drei PGE 2 -Syntheseenzymen in HUCMSCs schlagen wir vor, dass PGE 2 eine wichtige vorteilhafte Komponente von HUCMSC-CM mit vasodilatatorischen und regenerativen Eigenschaften ist PAH.Wir identifizierten bei unserem Patienten eine De-novo-Mißense-Mutation im ACVRL1 -Gen. Interessanterweise wurde diese spezielle ACVRL1 -Funktionsverlust-Mutation (c.1451 G>A, p.(Arg484Gln)) bei keinem Patienten mit HHT in Abwesenheit von PAH gefunden, was die Wirkung dieser Einzelnukleotid-Variante auf unterstreicht Lungengefäßentwicklung und Homöostase.Unsere Hauptabsicht war es, über die beeindruckenden Verbesserungen bei dem behandelten Kind und die wahrscheinlichen Mechanismen zu berichten, die durch unsere Multi-Omics-Analyse des Falls und die verfügbaren Nabelschnurproben aufgedeckt wurden. Hier demonstrieren wir die Sicherheit und Wirksamkeit einer von MSC abgeleiteten Therapie, jedoch nur bei einem Patienten. Zusätzliche Einschränkungen unserer Studie sind eine relativ kleine Anzahl von Validierungsfällen (drei Nabelschnüre). Weitere Studien sind für eine strenge Validierung unserer Ergebnisse erforderlich.Abschließend lässt sich sagen, dass serielle Infusionen von HUCMSC-CM bei einem jungen Patienten mit schwerer PAH zu einer deutlichen klinischen und hämodynamischen Verbesserung führten. Wir gehen davon aus, dass die von HUCMSC abgeleitete Therapie das Potenzial hat, eine wirksame Behandlung für die schwersten Formen der klinischen PAH zu werden. Weitere prospektive klinische Studien sind gerechtfertigt, um die Vorteile einer von HUCMSC abgeleiteten PAH-Therapie zu untersuchen.MethodenIsolierung von HUCMSC und HUVEC und Herstellung von Zell-abgeleitetem CMIsolierung, Kultur und Charakterisierung von primärem HUCMSC und HUVECMSC-ähnliche Zellen wurden aus der menschlichen Nabelschnur (Wharton's Jelly) nach der Geburt eines voll ausgetragenen (38. Schwangerschaftswoche) Säuglings (hier: jüngere Schwester, per Kaiserschnitt; Abb.
1
und
2
) und aus zusätzlicher menschlicher Nabelschnur isoliert Schnüre (Abb.
3
). Die Zellen wurden durch Explantatkultur in MSC-Wachstumsmedium
40
kultiviert . Kurz gesagt wurde Nabelschnurgewebe mehrere Male mit PBS gewaschen, um Blutzellen zu entfernen, in etwa 1,5 cm 3 große Stücke geschnitten und in MSC-Wachstumsmedium (&agr;MEM, Invitrogen), ergänzt mit 15 % allogenem humanem AB-Serum (HS ), 100 U ml –1 Penicillin, 100 mg ml –1von Streptomycin und 2 mM L-Glutamin bei 37°C in einer befeuchteten Atmosphäre mit 5% CO 2 . Die Explantatkultur wurde 15 Tage lang durchgeführt. Der Auswuchs einer adhärenten angereicherten MSC-Population wurde durch Behandlung mit Accutase (Capricorn Scientific) gemäß dem Protokoll des Herstellers für 5 Minuten bei 37 °C geerntet. Die Zellen wurden bei 320 g für 5 Minuten zentrifugiert, in MSC-Kulturmedium (&agr;MEM), ergänzt mit 10 % HS, 100 U ml –1 Penicillin, 100 mg ml –1 Streptomycin und 2 mM L-Glutamin, bei 37° resuspendiert C in einer befeuchteten Atmosphäre mit 5 % CO 2 und bei einer Dichte von 4.000 Zellen pro cm 2 kultiviert. Das Ernten und die Subkultivierung in entsprechenden Passagen erfolgte nach Behandlung mit Accutase (Capricorn Scientific) bei 37°C für 3 Minuten.Kontinuierlich proliferierende MSCs wurden geerntet und auf Zellzyklusprogression und Zelloberflächenmarkerexpression durch Durchflusszytometrie
40
analysiert . Neben nachweisbaren G1-, S- und G2/M-Phasen wurde das Vorhandensein von CD73, CD90 und CD105 bei gleichzeitiger Abwesenheit von CD14, CD31, CD34, CD45 und HLA-DR durch FACS-Analyse gemäß dem Vorschlag der International Society for Cellular getestet Therapie als eines der Minimalkriterien für die MSC-Charakterisierung
41
. HUVECs wurden von PromoCell (Kat.-Nr. C-12200, Heidelberg, Deutschland) bezogen und gemäß den Anweisungen des Herstellers (PromoCell Instruction Manual) bis P3 oder P6 (scRNA-seq) subkultiviert. Für Proteom- und LC-MS-Analysen wurde CM von HUCMSCs und HUVECs nach 3 Waschschritten in geeignetem serumfreiem Medium und Inkubation der Kulturen in entsprechendem serumfreiem Medium für 36 h gesammelt, gefolgt von Zentrifugation (3.185 g für 10 Minuten). .Herstellung von Zell-abgeleitetem CM aus HUCMSC zur intravaskulären InfusionNach der MSC-Kultur in den Passagen 2 und 3 in der subkonfluenten Wachstumsphase wurde der serumfreie Überstand als CM nach 36 Stunden geerntet, zentrifugiert (3,185 gfür 10 Minuten), negativ auf Bakterien- und Mykoplasmenkontamination getestet und bei −80 °C kryokonserviert. In der Nacht vor der Injektion (etwa 12–14 Stunden) wurde das HUCMSC-CM vorsichtig bei 4 °C aufgetaut und dann auf Raumtemperatur vorgewärmt. Für die HUCMSC-CM-Infusionen wurden bestimmte Filter verwendet: Zeit 0, CATH #1 (ipa): Dosis 1, 200 ml CM, Sterifix 0,2-μm-Filter (B. Braun, 4099303); Zeitpunkt 1, CATH #2 Dosis 2, ipa: 200 ml CM, Transfusionsfilter 200 µm (B. Braun, 8270066SP). Zeit 1, Dosis 3, SVC; 200 ml KM, Transfusionsfilter 200 µm (B. Braun, 8270066SP). Zeitpunkt 1, Dosis 4, SVC: 200 ml KM, Minisart 0,2 µm Filter (Sartorius/Th. Geyer, 90491011), Transfusionsfilter 200 µm (B. Braun). Zeitpunkt 1, Dosis 5, SVC: 200 ml KM, Minisart 0,2 µm Filter (Sartorius/Th. Geyer, 90491011), Transfusionsfilter 200 µm (B. Braun).Herzkatheterisierungen und Infusionen von HUCMSC-CMNach der Diagnose (CATH #0) wurden alle nachfolgenden Herzkatheteruntersuchungen an der Medizinischen Hochschule Hannover im Dezember 2019 (Zeitpunkt 0, CATH #1), Februar 2020 (Zeitpunkt 1, Cath #2) und Mai 2020 (Zeitpunkt 2, CATH #3) durchgeführt ) (Abb.
1a
und Ergänzungstabelle
1
). Die PAH-gerichtete Medikation wurde in den 6 Monaten vor CATH #1, als zum ersten Mal HUCMSC-CM in den PAs verabreicht wurde, nicht geändert und danach nicht geändert.Während CATH Nr. 1 und CATH Nr. 2 wurden nach vollständiger invasiver hämodynamischer Beurteilung 200 ml HUCMSC-CM in die PAs (ipa) über 1 Stunde infundiert – d. h. 100 ml in die rechte Pulmonalarterie (RPA) über 30 Minuten und 100 ml in die linke Pulmonalarterie (LPA) über 30 Minuten. In der Woche nach CATH Nr. 2 wurden 200 ml HUCMSC-CM über einen zentralvenösen Zugang über 60 Minuten an den Tagen 1, 2 und 3 nach CATH Nr. 2 infundiert (Abb.
1a
und ergänzende Abb.
6
).Mikrobiologische und immunologische Tests von HUCMSC, HUCMSC-CM, Empfänger (Patient), Spender (Schwester) und MutterHUCMSC/HUCMSC-CM (Zellkulturüberstand)Kultur 10–14 Tage negativ für Bakterienwachstum (aerob und anaerob)SpenderHLA-Typen:DNA-Typen HLA-1: A*30, A*68, B*13, B*18, C*06, C*12DNA-Typen HLA-2: DRB3*pos., DQB1*03HBs-Antigen-negativ, Anti-HBC-negativ, Anti-HBS 28 IE l –1Anti-HCV-negativ, CMV-IgM-negativ, CMV-IgG 606 U ml −1EBV-IgG 571 E ml –1 , VCA IgM negativ, EBNA1-IgG 141 E ml –1Toxoplasma-Suchtest negativTreponema pallidum IA-Test negativHIV-AK1/2, p24-Ag-negativEmpfängerBlutgruppe 0 Rh positiv, CcDD.Ee, K-, unregelmäßige Erythrozyten-Antikörper negativHLA-Typen:DNA-Typen HLA-1: A*24, A*68, B*13, B*18, C*06, C*12DNA-Typen HLA-2: DRB1*07, DRB1*11, DRB3*pos., DRB4*pos., DQB1*02, DQB1*03GvH-Konstellation (MSC-Transplantat versus Patient): A*24, C*04, DRB1*07, DQB1*02HvG-Konstellation (Patient gegen MSC-Transplantation): A*68, C*06Mycoplasma-IgM-negativ, Mycoplasma-IgG-negativDiagnosekriterien von Curaçao für HHT (Osler-Weber-Rendu-Syndrom) gemäß internationalen Richtlinien (2000 und 2020)
42
,
43
Zu diesen Kriterien gehören
[list=none]
[*](1)wiederkehrende und spontane Epistaxis;
[*](2)das Vorhandensein mehrerer mukokutaner Teleangiektasien (charakteristische Stellen: Lippen, Mundhöhle, Finger und Nase);
[*](3)viszerale Lokalisation von Läsionen (wie gastrointestinale Teleangiektasien und pulmonale, hepatische, zerebrale oder spinale arteriovenöse Missbildungen);
[*](4)ein betroffenes Familienmitglied ersten Grades mit HHT gemäß diesen Kriterien. Wenn eine Person drei oder mehr Kriterien aufweist, wird davon ausgegangen, dass sie HHT hat; wenn sie zwei Kriterien erfüllen, ist die Diagnose möglich; und mit einem oder keinem Kriterium wird HHT unter Verwendung dieser Kriterien als unwahrscheinlich betrachtet. Eine HHT-Diagnose gilt als „sicher“, wenn drei oder mehr Curaçao-Kriterien vorliegen, als „möglich oder vermutet“, wenn zwei Kriterien vorliegen, und als „unwahrscheinlich“, wenn ein oder kein Kriterium vorliegt.
[/list]
Einzelzellsequenzierung von MSC- und HUVEC-ZellprobenDie Bibliotheksvorbereitung für die Einzelzell-mRNA-Sequenzierungsanalyse wurde gemäß dem Chromium NextGEM Single Cell 3′ Reagent Kit Version 3.1 User Guide (Manual Part Number CG000204 Rev B, 10x Genomics) durchgeführt. Ein zweifacher Überschuss an Zellen wurde in dem angegebenen Volumen in den 10x-Controller geladen, um eine Zielzahl von 1.500 Zellen pro Probe zu erreichen. Gleiche molare Anteile von acht generierten Bibliotheken wurden entsprechend gepoolt, mit NaOH denaturiert und schließlich gemäß Denature and Dilute Libraries Guide (Dokument 15048776 v02, Illumina) auf 1,8 pM verdünnt. Als nächstes wurden 1,3 ml des denaturierten Pools auf einem Illumina NextSeq 550-Sequenzer unter Verwendung einer High Output Flowcell für 75 Zyklen und 400 Millionen Cluster (20024906, Illumina) sequenziert. Die proprietäre 10x Genomics CellRanger-Pipeline (Version 4.0. 0) wurde mit Standardparametern verwendet, mit Ausnahme der Einstellung der erwarteten Zellen (–expect-cells 1500). CellRanger wurde verwendet, um gelesene Daten mit dem von 10x Genomics bereitgestellten menschlichen Referenzgenom (refdata-gex-GRCh38-2020-A) unter Verwendung des STAR-Aligners abzugleichen. Die durchschnittliche Anzahl der Reads pro Zelle lag bei allen Proben zwischen 29.916 und 42.827. Die mittlere Anzahl von Genen pro Zelle lag in allen Proben zwischen 3.578 und 4.465.ELISAEDTA-Blut wurde während der Herzkatheterisierung aus der oberen Hohlvene (SVC) entnommen. Die Blutproben wurden sofort für 10 Minuten bei 1.300 g zentrifugiert. Das Plasma wurde aliquotiert und bei –80 °C gelagert. Plasmaproben wurden 1:3 mit Probenverdünnungsmittel verdünnt, und die Plasmakonzentrationen von neuralem Vorläuferzellen-exprimiertem entwicklungsmäßig herunterreguliertem Protein 9 (NEDD9) wurden gemäß den Anweisungen des Herstellers bestimmt (Aviva Systems Biology, OKEH02459, Lot KE0777). Kurz gesagt, 100 µl Standards, verdünnte Proben und Blindprobe wurden in die Vertiefungen der NEDD9-Mikrotiterplatte gegeben und 2 Stunden lang bei 37 °C inkubiert. Die Flüssigkeit wurde verworfen und 100 µl biotinylierter NEDD9-Erkennungsantikörper wurden in jede Vertiefung gegeben und 60 Minuten lang bei 37 °C inkubiert. Flüssigkeit wurde entfernt und die Mikroplatte wurde mit Waschpuffer gewaschen. Dann wurden 100 µl Avidin-HRP-Konjugat in jede Vertiefung gegeben und 60 Minuten bei 37 °C inkubiert, gefolgt von einem weiteren Waschschritt. Nächste, 50 µl TMB-Substrat wurden hinzugefügt und 15 Minuten lang bei 37 °C im Dunkeln inkubiert. Schließlich wurden 50 µl Stopplösung in jede Vertiefung gegeben und die Extinktion bei 450 nm mit einer Wellenlängenkorrektur von 570 nm abgelesen.Plasmakonzentrationen von ICAM-1 (Probenverdünnung 1:1.000), SAA (Probenverdünnung 1:1.000) und IFN-γ (Probenverdünnung 1:2) wurden unter Anwendung der Multi-Array-Technologie von Meso Scale Discovery gemäß den Anweisungen des Herstellers gemessen. ICAM-1 und SAA wurden im Vascular Injury Panel (K15198D-1) gemessen, und IFN-γ wurde im Proinflammatory Panel (K15049D-1) nachgewiesen, beides gemäß den Anweisungen des Herstellers. Kurz gesagt, die Platten wurden dreimal mit 150 µl Waschpuffer pro Vertiefung gewaschen, und 25 µl verdünnte Probe, Kalibrator oder Kontrolle wurden pro Vertiefung (für ICAM-1 und SAA) oder 50 µl verdünnte Probe hinzugefügt. Kalibrator oder Kontrolle für IFN-γ. Die Platten wurden dann bei Raumtemperatur für 2 Stunden unter Schütteln inkubiert. Nach der Inkubation wurden die Platten dreimal mit 150 &mgr;l Waschpuffer pro Vertiefung gewaschen; 25 ul Detektionsantikörper wurden in jede Vertiefung gegeben; und die Platte wurde für eine weitere Stunde (für ICAM-1 und SAA) oder 2 Stunden für IFN-γ unter Schütteln bei Raumtemperatur inkubiert. Schließlich wurden die Platten dreimal mit 150 &mgr;l Waschpuffer pro Vertiefung gewaschen, und 150 &mgr;l 1 × Lesepuffer wurden pro Vertiefung (für ICAM-1 und SAA) oder 2 × Lesepuffer für IFN-&ggr; hinzugefügt. Signalintensitäten wurden mit einem MESO QuickPlex SQ 120-Instrument und der Discovery Workbench-Software, Version 4.0 (Meso Scale Discovery), erfasst und analysiert. Die durchschnittlichen Proteinkonzentrationen aus der oberen Hohlvene (SVC) und der unteren Hohlvene (IVC) und dem rechten Vorhof (RA) werden angegeben. die Platten wurden dreimal mit 150 &mgr;l Waschpuffer pro Vertiefung gewaschen, und 150 &mgr;l 1 × Lesepuffer wurden pro Vertiefung (für ICAM-1 und SAA) oder 2 × Lesepuffer für IFN-&ggr; hinzugefügt. Signalintensitäten wurden mit einem MESO QuickPlex SQ 120-Instrument und der Discovery Workbench-Software, Version 4.0 (Meso Scale Discovery), erfasst und analysiert. Die durchschnittlichen Proteinkonzentrationen aus der oberen Hohlvene (SVC) und der unteren Hohlvene (IVC) und dem rechten Vorhof (RA) werden angegeben. die Platten wurden dreimal mit 150 &mgr;l Waschpuffer pro Vertiefung gewaschen, und 150 &mgr;l 1 × Lesepuffer wurden pro Vertiefung (für ICAM-1 und SAA) oder 2 × Lesepuffer für IFN-&ggr; hinzugefügt. Signalintensitäten wurden mit einem MESO QuickPlex SQ 120-Instrument und der Discovery Workbench-Software, Version 4.0 (Meso Scale Discovery), erfasst und analysiert. Die durchschnittlichen Proteinkonzentrationen aus der oberen Hohlvene (SVC) und der unteren Hohlvene (IVC) und dem rechten Vorhof (RA) werden angegeben.Markierungsfreie quantitative EntdeckungsproteomikProbenvorbereitung für die ProteomikProtein wurde aus HUVECs und HUCMSCs extrahiert, und DNA wurde in 40 &mgr;l Lysepuffer (1 % SDS, 0,1 M ABC, 1,25 × PIC) in AFA-TUBE TPX Strips auf einem Covaris LE220Rsc durch fokussierte Ultraschallbehandlung (PIP 450 W, DF 25 %, CPB 200, zwei Wiederholungen, 300-Sekunden-Impuls, 20 °C). Die Proben wurden von Trümmern befreit (2.500 g für 5 Minuten) und das Protein quantifiziert (Pierce BCA, 23225). Proben von 30 &mgr;g Zellprotein wurden mit Lysepuffer auf 50 &mgr;l aufgefüllt, und 16,6 &mgr;l Reduktions- und Alkylierungspuffer (40 mM TCEP, 160 mM CAA, 200 mM ABC) wurden hinzugefügt. Sekretierte Proteine im CM (200 &mgr;l) wurden konzentriert (Gefriertrocknung über Nacht) und in 40 &mgr;l 10 mM TCEP und 40 mM CAA rekonstituiert. Zelluläre und sezernierte Proteine wurden unter Verwendung des SP3-Protokolls mit einstufiger Reduktion und Alkylierung 44 hergestellt auf einer Beckman Biomek i7 Workstation. Die Proben wurden für 5 Minuten bei 95°C inkubiert und auf Raumtemperatur abgekühlt. Proteine wurden an 250-μg-paramagnetische Kügelchen gebunden (1:1-Verhältnis von hydrophilen/hydrophoben Kügelchen) durch Zugabe von Acetonitril (ACN) zu 50 % für zelluläre Proteine bzw. 70 % für sezernierte Proteine. Die Proben wurden zweimal mit 80 % Ethanol und einmal mit 100 % ACN gewaschen, bevor sie in 35 &mgr;l 100 mM ABC rekonstituiert wurden. Der Verdau wurde über Nacht bei 37 °C unter Verwendung einer Trypsin/LysC-Enzymmischung (Promega) bei einem Verhältnis von Protein:Enzym von 50:1 für zelluläre Proteine bzw. 250 ng für sezernierte Proteine abgeschlossen. Die Reaktion wurde mit FA (0,1 %) gestoppt, und die Peptide wurden bis zur Analyse ohne weitere Konditionierung oder Reinigung bei −80 °C gelagert.Proteomanalyse durch datenunabhängige Akquisition LC–MSDie Menge des injizierten tryptischen Verdaus wurde auf 40 ng eingestellt, das verfügbare Material für die niedrigst konzentrierte Probe. Die Peptide wurden auf einer 25-cm-Aurora-Serie mit Emittersäule (CSI, 25 cm × 75 µm ID, 1,6 µm C18, IonOpticks, installiert in der Nano-Elektrospray-Quelle (CaptiveSpray-Quelle, Bruker Daltonics) bei 50 °C unter Verwendung von aufgelöst UltiMate 3000 (Thermo Fisher Scientific Dionex) gekoppelt mit TIMS-Quadrupol-Flugzeitinstrument (timsTOF Pro2, Bruker Daltonics) und gemessen im diaPASEF-Modus Die mobilen Phasen Wasser/0,1 % FA und ACN/0,1 % FA (A und B, jeweils) wurden in den linearen Gradienten angewendet, beginnend mit 2 % B und ansteigend auf 17 % in 87 Minuten, gefolgt von einer Erhöhung auf 25 % B in 93 Minuten, 37 % B in 98 Minuten und 80 % B in 99–104 Minuten, und die Säule wurde in den nächsten 15 Minuten in 2 % B äquilibriert.m / z [Th], 1/ K0 [Th]: 622,0289 , 0,9848; 922.0097, 1.1895; 1221.9906, 1.3820). Das diaPASEF-Fensterschema reichte in der Dimension m / z von 396 bis 1.103 Th und in der Dimension 1/ K 0 0,7–1,3 Vs cm-2, mit 59 × 12 Th-Fenstern). Alle Messungen wurden im Modus „Low Sample Amount“ mit einer Rampenzeit von 166 ms durchgeführt.Proteinidentifizierung und -quantifizierungDie Rohdaten wurden mit DIA-NN 1.8 (Ref. 45 ) mit dem Ionenmobilitätsmodul für diaPASEF verarbeitet (Demichev et al., www.biorxiv.org/content/10.1101/2021.03.08.434385v1 (2021)) . Die Massengenauigkeiten von MS2 und MS1 wurden beide auf 10 ppm eingestellt, und die Scanfenstergröße wurde auf 10 eingestellt. DIA-NN wurde im bibliotheksfreien Modus mit Standardeinstellungen (Fasta Digest und Deep-Learning-basierte Spektren, RT- und IMs-Vorhersage) ausgeführt. unter Verwendung der UniProt Human Reference Proteome Annotations ( UP000005640_9606 , heruntergeladen am 20. Dezember 2019) 46 und der Match-Between-Runs (MBR)-Option.LC-MS von Zellen und CMDie Proben wurden mit internen Standards bis zu einer Endkonzentration von 1,0 ng ml −1 versetzt und mittels Festphasenextraktion wie zuvor beschrieben präpariert 47 , und 200 &mgr;l CM-Proben wurden verwendet. Mediumproben wurden in 200 &mgr;l 40% MeOH und Zellpelletproben in 100 &mgr;l 40% MeOH rekonstituiert. Die LC-MS-Analyse wurde mit zwei LC-30AD-Pumpen, einem SIL-30AC-Autosampler und einem CTO-20AC-Säulenofen (alle Shimadzu) durchgeführt. Der Autosampler wurde bei 6 °C gehalten. Vierzig Mikroliter wurden injiziert, und die Trennung wurde auf einer Kinetex C18-Säule (Phenomenex, 50 × 2,1 mm, 1,7 &mgr;m) unter Verwendung eines Gradienten von 0,01 % Essigsäure (Fluka) in Wasser (Honeywell-Riedel de Haën; A) und 0,01 % Essigsäure in MeOH ( . Der Ofen wurde bei 50°C gehalten. Der Gradient war wie folgt: 0,0–1,0 Minuten konstant bei 30 % B, 1,0–1,1 Minuten linearer Anstieg auf 45 % B, 1,1–2,0 Minuten linearer Anstieg auf 53,5 % B, 2,0–4,4 Minuten linearer Anstieg auf 55,5 % B, 4,0 –7,0 Minuten linearer Anstieg auf 90 % B, 7,0–7,1 Minuten linearer Anstieg auf 100 % B, 7,1–9. 0 Minuten konstant bei 100 % B, 9,0–9,5 Minuten lineare Abnahme auf 30 % B, 9,5–11,5 Minuten konstant bei 30 % B. Die Detektion erfolgte auf einem Qtrap 6500 (Sciex Nieuwerkerk a/d IJssel), ausgestattet mit einer ESI-Quelle. Das MS wurde im negativ geplanten MRM-Modus betrieben. Die Nadelspannung der Quelle wurde auf –4.500 V, die Trocknungstemperatur auf 450 °C, Ionenquellengas 1 und 2 (beide Luft) auf 40 psi bzw. 30 psi und das Zerstäubergas (Stickstoff) auf 30 psi eingestellt Das Eintrittspotential wurde auf 10 V und der Stoßgasfluss auf „mittel“ eingestellt. Detaillierte Einstellungen finden Sie an anderer Stelle Die Nadelspannung der Quelle wurde auf –4.500 V, die Trocknungstemperatur auf 450 °C, Ionenquellengas 1 und 2 (beide Luft) auf 40 psi bzw. 30 psi und das Zerstäubergas (Stickstoff) auf 30 psi eingestellt Das Eintrittspotential wurde auf 10 V und der Stoßgasfluss auf „mittel“ eingestellt. Detaillierte Einstellungen finden Sie an anderer Stelle Die Nadelspannung der Quelle wurde auf –4.500 V, die Trocknungstemperatur auf 450 °C, Ionenquellengas 1 und 2 (beide Luft) auf 40 psi bzw. 30 psi und das Zerstäubergas (Stickstoff) auf 30 psi eingestellt Das Eintrittspotential wurde auf 10 V und der Stoßgasfluss auf „mittel“ eingestellt. Detaillierte Einstellungen finden Sie an anderer Stelle
48
. Kalibrierbereiche und Funktionen sind in den Zusatztabellen
6
und
7
angegeben . Alle Eichlinien wurden 1/x 2 gewogen . Die Ergebnisse wurden als ng ml –1 oder ng Prostaglandin pro mg Protein (Zellpellet) ausgedrückt. Protein wurde unter Verwendung des BCA-Assays gemäß den Anweisungen des Herstellers quantifiziert.Um die regenerative, von MSC abgeleitete Wirkung von PGE 2 zu bestätigen , schlossen wir nur eine kleine Gruppe ausgewählter Prostaglandine (PGD 2 , PGF 2α , PGE 2 , 8-iso-PGE 2 und 8-iso-PGF 2α ) und deren Vorläufer AA ein in der LC–MS/MS-Analyse.statistische AnalyseDie scRNA-seq-Datenanalyse wurde unter Verwendung des Seurat R-Pakets (Version 4.0.2) durchgeführt. Es wurden zwei Arten von scRNA-seq-Analysen durchgeführt. (1) Die HUCMSCs, die das zur Behandlung des gemeldeten Falls verwendete CM erzeugten (Proben-ID: HUCMSC1_P3_female; Zellnummer: 1418), wurden auf das Vorhandensein von Zellclustern mit Regenerationspotenzial analysiert. Nach den Standardanalyseschritten in Seurat (wie im Handbuch beschrieben), einschließlich der Regression der Zellzykluseffekte, führten wir ein unbeaufsichtigtes Clustering der Einzelzelldaten durch. (2) Integrierte scRNA-seq-Analyse von drei anderen HUCMSC-Proben (Proben-IDs: HUCMSC2_P6_männlich, HUCMSC3_P6_männlich, HUCMSC4_P6_männlich; jeweilige Zellnummern: 1317, 1982 und 3203) und zwei HUVEC-Proben (Proben-IDs: HUVEC1_P6_männlich, HUVEC2_P6_männlich; jeweilige Zellnummern: 1316 und 2397) wurde wie im Seurat-Tutorial (
satijalab.org/seurat/archive/v3.1/immune_alignment.html
). Vor dieser Analyse wurde eine Standardfiltration durchgeführt. Angesichts der Tatsache, dass der Unterschied in den Proliferationsraten zwischen HUCMSCs und HUVECs biologisch relevant sein kann, haben wir uns entschieden, die Zellzykluseffekte nicht zu regressieren. Die Stapeleffektkorrektur und -normalisierung wurde mit der SCTransform-Funktion aus dem SCTransform R-Paket (Version 0.3.3) durchgeführt. Der Prozentsatz der mitochondrialen Gene und Proben-IDs wurden als Variablen für die Regression in SCTransform verwendet. Nach der Batch-Effekt-Korrektur reichten die Stichprobenprozentsätze pro Cluster wie folgt: Cluster 0 (16,87–21,67 %), Cluster 1 (18,47–20,9 %), Cluster 2 (15,02–25,17 %), Cluster 3 (18,53–21,01 %), Cluster 4 (13,19–26,44 %), Cluster 5 (16,14–23,87 %), Cluster 6 (13,03–27,58 %) und Cluster 7 (0–40,44 %). Seurats Funktion SelectIntegrationFeatures mit der Anzahl der Features auf 3 gesetzt, 000 wurde für die Merkmalsauswahl verwendet. Die ElbowPlot-Funktion von Seurat wurde verwendet, um die Anzahl sinnvoller Dimensionen abzuschätzen. Die zur Definition der Zellhaufen verwendeten Markergene oder differentiell exprimierten Gene (False Discovery Rate (FDR)-adjustedP < 0,05) aus der integrierten Analyse (HUCMSC versus HUVEC) wurden mit dem Online-Tool Enrichr (
maayanlab.cloud/Enrichr/
) auf GO- und Signalweg-Überrepräsentation analysiert. Differentiell exprimierte Gene wurden unter Verwendung der FindMarkers-Funktion mit Standardparametern aus dem Seurat-Paket identifiziert.Die Proteomikanalyse wurde unter Verwendung des DEP R-Pakets (Version 1.12.0) mit Standardparametern durchgeführt. Darüber hinaus haben wir GSEA (Version 4.1.0) mit Standardparametern verwendet, um eine Genanreicherungssatzanalyse von differentiell exprimierten Genen und differentiell angereicherten Proteinen durchzuführen. Prostaglandin (LC-MS)-Ergebnisse wurden mit GraphPad Prism (Version 7) analysiert. Es wurden zweiseitige Mann-Whitney- U - Tests verwendet, da die Normalität aufgrund kleiner Stichprobenumfänge nicht überprüft werden konnte.Ethische ÜberlegungenDie Verwendung von primären humanen MSCs nach Explantatkultur aus Nabelschnurgewebe wurde von der Ethikkommission der Medizinischen Hochschule Hannover (Research Obstetric Biobank; Institutional Review Board Number 1303-2012/update 2020) genehmigt. Die Betreuer (Eltern) der behandelten Patientin gaben eine schriftliche Einverständniserklärung zur „Compassionate Use“-Therapie, Bioanalyse und Veröffentlichung der Daten. Dieser Bericht entspricht den CARE-Richtlinien.Zusammenfassung der BerichterstattungWeitere Informationen zum Forschungsdesign finden Sie in der mit diesem Artikel verlinkten
Nature Research Reporting Summary .
DatenverfügbarkeitDie scRNA-seq-Daten sind über das National Center of Biotechnology Information Gene Expression Omnibus (Zugangs-ID:
GSE199071
) zugänglich. Wir haben die Rohdaten für Proteomik-Experimente bei PRIDE (EMBL) hinterlegt, das Teil von ProteomeXchange ist (Zugangs-ID:
PXD032234
). Die LC-MS-Prostaglandindaten befinden sich im
. Der Ofen wurde bei 50°C gehalten. Der Gradient war wie folgt: 0,0–1,0 Minuten konstant bei 30 % B, 1,0–1,1 Minuten linearer Anstieg auf 45 % B, 1,1–2,0 Minuten linearer Anstieg auf 53,5 % B, 2,0–4,4 Minuten linearer Anstieg auf 55,5 % B, 4,0 –7,0 Minuten linearer Anstieg auf 90 % B, 7,0–7,1 Minuten linearer Anstieg auf 100 % B, 7,1–9. 0 Minuten konstant bei 100 % B, 9,0–9,5 Minuten lineare Abnahme auf 30 % B, 9,5–11,5 Minuten konstant bei 30 % B. Die Detektion erfolgte auf einem Qtrap 6500 (Sciex Nieuwerkerk a/d IJssel), ausgestattet mit einer ESI-Quelle. Das MS wurde im negativ geplanten MRM-Modus betrieben. Die Nadelspannung der Quelle wurde auf –4.500 V, die Trocknungstemperatur auf 450 °C, Ionenquellengas 1 und 2 (beide Luft) auf 40 psi bzw. 30 psi und das Zerstäubergas (Stickstoff) auf 30 psi eingestellt Das Eintrittspotential wurde auf 10 V und der Stoßgasfluss auf „mittel“ eingestellt. Detaillierte Einstellungen finden Sie an anderer Stelle Die Nadelspannung der Quelle wurde auf –4.500 V, die Trocknungstemperatur auf 450 °C, Ionenquellengas 1 und 2 (beide Luft) auf 40 psi bzw. 30 psi und das Zerstäubergas (Stickstoff) auf 30 psi eingestellt Das Eintrittspotential wurde auf 10 V und der Stoßgasfluss auf „mittel“ eingestellt. Detaillierte Einstellungen finden Sie an anderer Stelle Die Nadelspannung der Quelle wurde auf –4.500 V, die Trocknungstemperatur auf 450 °C, Ionenquellengas 1 und 2 (beide Luft) auf 40 psi bzw. 30 psi und das Zerstäubergas (Stickstoff) auf 30 psi eingestellt Das Eintrittspotential wurde auf 10 V und der Stoßgasfluss auf „mittel“ eingestellt. Detaillierte Einstellungen finden Sie an anderer Stelle
48
. Kalibrierbereiche und Funktionen sind in den Zusatztabellen
6
und
7
angegeben . Alle Eichlinien wurden 1/x 2 gewogen . Die Ergebnisse wurden als ng ml –1 oder ng Prostaglandin pro mg Protein (Zellpellet) ausgedrückt. Protein wurde unter Verwendung des BCA-Assays gemäß den Anweisungen des Herstellers quantifiziert.Um die regenerative, von MSC abgeleitete Wirkung von PGE 2 zu bestätigen , schlossen wir nur eine kleine Gruppe ausgewählter Prostaglandine (PGD 2 , PGF 2α , PGE 2 , 8-iso-PGE 2 und 8-iso-PGF 2α ) und deren Vorläufer AA ein in der LC–MS/MS-Analyse.statistische AnalyseDie scRNA-seq-Datenanalyse wurde unter Verwendung des Seurat R-Pakets (Version 4.0.2) durchgeführt. Es wurden zwei Arten von scRNA-seq-Analysen durchgeführt. (1) Die HUCMSCs, die das zur Behandlung des gemeldeten Falls verwendete CM erzeugten (Proben-ID: HUCMSC1_P3_female; Zellnummer: 1418), wurden auf das Vorhandensein von Zellclustern mit Regenerationspotenzial analysiert. Nach den Standardanalyseschritten in Seurat (wie im Handbuch beschrieben), einschließlich der Regression der Zellzykluseffekte, führten wir ein unbeaufsichtigtes Clustering der Einzelzelldaten durch. (2) Integrierte scRNA-seq-Analyse von drei anderen HUCMSC-Proben (Proben-IDs: HUCMSC2_P6_männlich, HUCMSC3_P6_männlich, HUCMSC4_P6_männlich; jeweilige Zellnummern: 1317, 1982 und 3203) und zwei HUVEC-Proben (Proben-IDs: HUVEC1_P6_männlich, HUVEC2_P6_männlich; jeweilige Zellnummern: 1316 und 2397) wurde wie im Seurat-Tutorial (
satijalab.org/seurat/archive/v3.1/immune_alignment.html
). Vor dieser Analyse wurde eine Standardfiltration durchgeführt. Angesichts der Tatsache, dass der Unterschied in den Proliferationsraten zwischen HUCMSCs und HUVECs biologisch relevant sein kann, haben wir uns entschieden, die Zellzykluseffekte nicht zu regressieren. Die Stapeleffektkorrektur und -normalisierung wurde mit der SCTransform-Funktion aus dem SCTransform R-Paket (Version 0.3.3) durchgeführt. Der Prozentsatz der mitochondrialen Gene und Proben-IDs wurden als Variablen für die Regression in SCTransform verwendet. Nach der Batch-Effekt-Korrektur reichten die Stichprobenprozentsätze pro Cluster wie folgt: Cluster 0 (16,87–21,67 %), Cluster 1 (18,47–20,9 %), Cluster 2 (15,02–25,17 %), Cluster 3 (18,53–21,01 %), Cluster 4 (13,19–26,44 %), Cluster 5 (16,14–23,87 %), Cluster 6 (13,03–27,58 %) und Cluster 7 (0–40,44 %). Seurats Funktion SelectIntegrationFeatures mit der Anzahl der Features auf 3 gesetzt, 000 wurde für die Merkmalsauswahl verwendet. Die ElbowPlot-Funktion von Seurat wurde verwendet, um die Anzahl sinnvoller Dimensionen abzuschätzen. Die zur Definition der Zellhaufen verwendeten Markergene oder differentiell exprimierten Gene (False Discovery Rate (FDR)-adjustedP < 0,05) aus der integrierten Analyse (HUCMSC versus HUVEC) wurden mit dem Online-Tool Enrichr (
maayanlab.cloud/Enrichr/
) auf GO- und Signalweg-Überrepräsentation analysiert. Differentiell exprimierte Gene wurden unter Verwendung der FindMarkers-Funktion mit Standardparametern aus dem Seurat-Paket identifiziert.Die Proteomikanalyse wurde unter Verwendung des DEP R-Pakets (Version 1.12.0) mit Standardparametern durchgeführt. Darüber hinaus haben wir GSEA (Version 4.1.0) mit Standardparametern verwendet, um eine Genanreicherungssatzanalyse von differentiell exprimierten Genen und differentiell angereicherten Proteinen durchzuführen. Prostaglandin (LC-MS)-Ergebnisse wurden mit GraphPad Prism (Version 7) analysiert. Es wurden zweiseitige Mann-Whitney- U - Tests verwendet, da die Normalität aufgrund kleiner Stichprobenumfänge nicht überprüft werden konnte.Ethische ÜberlegungenDie Verwendung von primären humanen MSCs nach Explantatkultur aus Nabelschnurgewebe wurde von der Ethikkommission der Medizinischen Hochschule Hannover (Research Obstetric Biobank; Institutional Review Board Number 1303-2012/update 2020) genehmigt. Die Betreuer (Eltern) der behandelten Patientin gaben eine schriftliche Einverständniserklärung zur „Compassionate Use“-Therapie, Bioanalyse und Veröffentlichung der Daten. Dieser Bericht entspricht den CARE-Richtlinien.Zusammenfassung der BerichterstattungWeitere Informationen zum Forschungsdesign finden Sie in der mit diesem Artikel verlinkten
Nature Research Reporting Summary .
DatenverfügbarkeitDie scRNA-seq-Daten sind über das National Center of Biotechnology Information Gene Expression Omnibus (Zugangs-ID:
GSE199071
) zugänglich. Wir haben die Rohdaten für Proteomik-Experimente bei PRIDE (EMBL) hinterlegt, das Teil von ProteomeXchange ist (Zugangs-ID:
PXD032234
). Die LC-MS-Prostaglandindaten befinden sich im
www.nature.com/articles/s44161-022-00083...gyI9sDcQAbstraktHier berichten wir über die Anwendung einer humanen Nabelschnur-Mesenchym-Stammzelle (HUCMSC)-abgeleitete Therapie für pulmonale arterielle Hypertonie (PAH). Eine 3-jährige Frau stellte sich mit vererbbarer PAH in Verbindung mit erblicher hämorrhagischer Teleangiektasie vor und wurde 6 Monate lang mit seriellen intravaskulären Infusionen von konditionierten Medien (CM) aus allogenen HUCMSCs behandelt. Die Behandlung verbesserte die klinischen und hämodynamischen Parameter deutlich und verringerte Blutplasmamarker für Gefäßfibrose, Verletzungen und Entzündungen. Eine vergleichende Analyse von Einzelzell-RNA-Sequenzierungsdaten, die von drei HUCMSCs und zwei humanen Nabelvenen-Endothelzellen (HUVEC)-Kontrollen gesammelt wurden, identifizierte acht gemeinsame Zellcluster, die alle auf ein für HUCMSCs spezifisches regeneratives Potenzial hindeuteten. Die Eigenschaften von HUCMSCs wurden durch nicht zielgerichtete, markierungsfreie Quantifizierung des Zell- und CM-Proteoms validiert, was auf eine erhöhte Aktivität der Regeneration, Autophagie und entzündungshemmender Wege und der mitochondrialen Funktion hindeutet. Die Prostaglandinanalyse zeigte eine erhöhte HUCMSC-Sekretion von Prostaglandin E2, das für seine Regenerationsfähigkeit bekannt ist. Zusätzliche prospektive klinische Studien sind gerechtfertigt, um die Vorteile einer von HUCMSC abgeleiteten Therapie für PAH zu bestätigen und weiter zu untersuchen.HauptsächlichPAH ist gekennzeichnet durch fortschreitenden, obliterativen Umbau der Lungenarteriolen, präkapillären Gefäßverlust, Rechtsherzinsuffizienz und Tod. Die Pathobiologie von Lungengefäßerkrankungen (PVD) und PAH ist komplex, multifaktoriell und wird durch Entzündungen und Stoffwechselstörungen vorangetrieben 1 . Trotz bemerkenswerter Verbesserungen in der Pharmakotherapie 2 , 3 ist fortgeschrittene PAH immer noch eine nicht heilbare, schwächende und tödliche Erkrankung 4 . Stamm- und Vorläuferzellen und/oder ihre sekretierten Produkte können effizientere Therapien für PAH darstellen 5 , 6 . Mitglieder der Transforming Growth Factor Beta (TGFβ)-Rezeptor-Superfamilie (Bone Morphogenetic Protein Receptor 2, BMPR2; Aktivin-A-Rezeptor-ähnlicher Typ 1, ACVRL1 ; und Endoglin, ENG ) und ihre Liganden spielen eine entscheidende Rolle in der Ätiologie von PAH 1 , 7 , 8 , 9 . Heterozygote Loss-of-Function-Mutationen in den Genen BMPR2 , ACVRL1 und ENG wurden unter anderem bei familiärer/heritabler PAH (HPAH) und idiopathischer PAH (IPAH) beschrieben 9 ; solche Mutationen finden sich auch bei hereditärer hämorrhagischer Teleangiektasie (HHT; Osler-Weber-Rendu-Krankheit). Patienten mit ACVRL1- Mutationen, die PAH entwickeln 7 sind besonders jung, haben oft einen raschen Krankheitsverlauf und eine schlechtere Prognose als Patienten mit BMPR2- Mutationen 10 . Hier demonstrieren wir eine sichere und effiziente HUCMSC-abgeleitete Behandlung schwerer, fortschreitender PAH mittels serieller intravaskulärer Infusionen von HUCMSC-CM bei einem jungen Patienten mit vererbbarer PAH und HHT Typ 2, verursacht durch eine ACVLR1 - Missense-Mutation.ErgebnisseZum Zeitpunkt der Diagnose befand sich das 3-jährige Mädchen in einem kritischen Zustand, nach zwei synkopalen „afebrilen Anfallsepisoden“, in Funktionsklasse 4 der Weltgesundheitsorganisation (WHO), mit einer 6-minütigen Gehstrecke (6MWD) von nur 270 Metern (SpO 2 > 95 %) und mäßige Thrombozytopenie bei 8 × 10 3 µl. Sie litt seit 10 Monaten unter Müdigkeit, wiederholtem Nasenbluten (Epistaxis) und mukokutanen Teleangiektasien an den Lippen, der Brust und den unteren Extremitäten. Serum-NTproBNP war mit 2.414 ng l –1 stark erhöht. Die Echokardiographie zeigte eine stark beeinträchtigte rechtsventrikuläre systolische Funktion (systolische Exkursion der Trikuspidalringebene (TAPSE), 1,4 cm) und Trikuspidalinsuffizienz, Grad 2. Die erste diagnostische Herzkatheterisierung (CATH #0; behandlungsnaiv) wurde im Januar 2019 an einem externen Tertiär durchgeführt Center. Invasive hämodynamische Messungen zeigten schwere suprasystemische PAH (Druck: Pulmonalarterie (PA), 119/57/85 mmHg; aufsteigende Aorta (AAO), 94/43/63 mmHg; mittlerer pulmonalarterieller Druck (mPAP)/mittlerer systemischer arterieller Druck (mSAP ) Ratio 1,35), stark erhöhter pulmonalvaskulärer Widerstand (PVRi, 21 WU × m²; PVR/SVR Ratio 1,2), Mangel an akuter Vasoreaktivität (AVT) und normaler Herzindex (Qsi, 3,6 l min −1 m −2). Dementsprechend erreichte der Patient bei der Diagnose eine „höhere Risiko“-Stratifizierung mit einem höheren Risiko-Score des European Pediatric PVD Network (EPPVDN) von bis zu 0,8 (12/15) und einem niedrigeren Risiko-Score von bis zu 0 (ergänzende Abb. 1 ). Das Elektrokardiogramm stimmte mit tachykardem Sinusrhythmus, rechtsatrialer Dilatation, RV-Hypertrophie und Belastung überein (ergänzende Abb. 2 ). Thoraxröntgen und Computertomographie (CT) zeigten eine starke Dilatation der RV und PAs, aber ein normales Lungenparenchym und keine Hinweise auf Thromben oder Venenverschlusserkrankungen (ergänzende Abb. 3 und 4 ). Pulmonale Angiogramme zeigten ein abnormes peripheres pulmonales Gefäßmuster, das durch eine sehr markante arterielle Schlängelung und Trübung des Kontrastmittels im peripheren Lungenkreislauf gekennzeichnet war; Letzteres kann durchgehend eine diffuse (prä)kapilläre Teleangiektasie und/oder sehr kleine arteriovenöse Fehlbildungen darstellen (ergänzende Abb. 5 und ergänzendes Video 1a,b ).Bei der Patientin wurde eine heterozygote Missense-Mutation im ACVRL1 -Gen festgestellt (c.1451 G>A, p.(Arg484Gln)). Diese Variante trat de novo auf und wurde zuvor bei sieben Patienten mit entweder isolierter PAH oder PAH plus HHT berichtet 11 , 12 , 13 , was sie dazu qualifiziert, vererbbare PAH und HHT Typ 2 zu haben.Nach der Diagnose (CATH #0) wurde der Patient mit einer dualen oralen Kombinationstherapie (Sildenafil und Bosentan) begonnen und zur Beurteilung der Lungentransplantation an die Medizinische Hochschule Hannover überwiesen 14 , 15 . Nach begrenztem Ansprechen auf die initiale duale orale Therapie wurde die gegen PAH gerichtete Pharmakotherapie modifiziert und umfasste orales Sildenafil, Macitentan, Spironolacton und inhalatives Iloprost. Da der systemische Blutdruck niedrig blieb (systolisch 80 mmHg), begannen wir nicht mit intravenösen Prostacyclin-Analoga. Unter diesen Maßnahmen wurde der Patient klinisch stabilisiert und die WHO-Funktionsklasse und 6MGT verbesserten sich (Ergänzungstabelle 1 ).Nichtsdestotrotz waren die echokardiographischen Variablen der Wechselwirkung zwischen rechtem Ventrikel und linkem Ventrikel (RV-LV) und der LV-Unterfüllung (RV/LV-endsystolisches Verhältnis, LV-endsystolischer Exzentrizitätsindex) und der Beschleunigungszeit der Pulmonalarterie (ein inverses Surrogat für den Schweregrad der PAH) unverändert stark unnormal. Die globale und longitudinale systolische RV-Funktion war reduziert. Aufgrund der düsteren Prognose wurde eine allogene HUCMSC-abgeleitete Therapie (Consented Compassionate Use) verfolgt.Dazu isolierten wir HUCMSCs 16 aus der Nabelschnur des jüngeren Geschwisters des Patienten und sammelten CM ( Methoden ). Der PAH-Patient erhielt eine Serie von fünf nicht GMP-zertifizierten, allogenen HUCMSC-CM-Infusionen über 6 Monate während zweier Krankenhausaufenthalte (Zeit 0 und Zeit 1) über einen intrapulmonalen Arterienkatheter und einen zentralen Venenkatheter. Alle HUCMSC-CM-Infusionen wurden sehr gut vertragen. Die Serum-CRP- und IL-6-Spiegel blieben normal, und der Patient erhielt keine antibiotischen, antiallergischen oder entzündungshemmenden Medikamente. Die Überwachung nach der Infusion erfolgte mindestens 24 Stunden, gefolgt von einer engmaschigen ambulanten Betreuung alle 1–6 Wochen. Der klinische Zustand und die invasive Hämodynamik wurden zu Studienbeginn (Zeitpunkt 0, CATH Nr. 1), nach 2 Monaten (Zeitpunkt 1, CATH Nr. 2) und nach 6 Monaten (Zeitpunkt 2, CATH Nr. 3) beurteilt (Abb. 1a ).Abb. 1: Die Behandlung mit HUCMSC-CM führt zu einer Verbesserung des Wachstums, der funktionellen Kapazität, der Risikobewertung und mehrerer hämodynamischer Variablen.



[list=none]
[*](1)wiederkehrende und spontane Epistaxis;
[*](2)das Vorhandensein mehrerer mukokutaner Teleangiektasien (charakteristische Stellen: Lippen, Mundhöhle, Finger und Nase);
[*](3)viszerale Lokalisation von Läsionen (wie gastrointestinale Teleangiektasien und pulmonale, hepatische, zerebrale oder spinale arteriovenöse Missbildungen);
[*](4)ein betroffenes Familienmitglied ersten Grades mit HHT gemäß diesen Kriterien. Wenn eine Person drei oder mehr Kriterien aufweist, wird davon ausgegangen, dass sie HHT hat; wenn sie zwei Kriterien erfüllen, ist die Diagnose möglich; und mit einem oder keinem Kriterium wird HHT unter Verwendung dieser Kriterien als unwahrscheinlich betrachtet. Eine HHT-Diagnose gilt als „sicher“, wenn drei oder mehr Curaçao-Kriterien vorliegen, als „möglich oder vermutet“, wenn zwei Kriterien vorliegen, und als „unwahrscheinlich“, wenn ein oder kein Kriterium vorliegt.
[/list]
Einzelzellsequenzierung von MSC- und HUVEC-ZellprobenDie Bibliotheksvorbereitung für die Einzelzell-mRNA-Sequenzierungsanalyse wurde gemäß dem Chromium NextGEM Single Cell 3′ Reagent Kit Version 3.1 User Guide (Manual Part Number CG000204 Rev B, 10x Genomics) durchgeführt. Ein zweifacher Überschuss an Zellen wurde in dem angegebenen Volumen in den 10x-Controller geladen, um eine Zielzahl von 1.500 Zellen pro Probe zu erreichen. Gleiche molare Anteile von acht generierten Bibliotheken wurden entsprechend gepoolt, mit NaOH denaturiert und schließlich gemäß Denature and Dilute Libraries Guide (Dokument 15048776 v02, Illumina) auf 1,8 pM verdünnt. Als nächstes wurden 1,3 ml des denaturierten Pools auf einem Illumina NextSeq 550-Sequenzer unter Verwendung einer High Output Flowcell für 75 Zyklen und 400 Millionen Cluster (20024906, Illumina) sequenziert. Die proprietäre 10x Genomics CellRanger-Pipeline (Version 4.0. 0) wurde mit Standardparametern verwendet, mit Ausnahme der Einstellung der erwarteten Zellen (–expect-cells 1500). CellRanger wurde verwendet, um gelesene Daten mit dem von 10x Genomics bereitgestellten menschlichen Referenzgenom (refdata-gex-GRCh38-2020-A) unter Verwendung des STAR-Aligners abzugleichen. Die durchschnittliche Anzahl der Reads pro Zelle lag bei allen Proben zwischen 29.916 und 42.827. Die mittlere Anzahl von Genen pro Zelle lag in allen Proben zwischen 3.578 und 4.465.ELISAEDTA-Blut wurde während der Herzkatheterisierung aus der oberen Hohlvene (SVC) entnommen. Die Blutproben wurden sofort für 10 Minuten bei 1.300 g zentrifugiert. Das Plasma wurde aliquotiert und bei –80 °C gelagert. Plasmaproben wurden 1:3 mit Probenverdünnungsmittel verdünnt, und die Plasmakonzentrationen von neuralem Vorläuferzellen-exprimiertem entwicklungsmäßig herunterreguliertem Protein 9 (NEDD9) wurden gemäß den Anweisungen des Herstellers bestimmt (Aviva Systems Biology, OKEH02459, Lot KE0777). Kurz gesagt, 100 µl Standards, verdünnte Proben und Blindprobe wurden in die Vertiefungen der NEDD9-Mikrotiterplatte gegeben und 2 Stunden lang bei 37 °C inkubiert. Die Flüssigkeit wurde verworfen und 100 µl biotinylierter NEDD9-Erkennungsantikörper wurden in jede Vertiefung gegeben und 60 Minuten lang bei 37 °C inkubiert. Flüssigkeit wurde entfernt und die Mikroplatte wurde mit Waschpuffer gewaschen. Dann wurden 100 µl Avidin-HRP-Konjugat in jede Vertiefung gegeben und 60 Minuten bei 37 °C inkubiert, gefolgt von einem weiteren Waschschritt. Nächste, 50 µl TMB-Substrat wurden hinzugefügt und 15 Minuten lang bei 37 °C im Dunkeln inkubiert. Schließlich wurden 50 µl Stopplösung in jede Vertiefung gegeben und die Extinktion bei 450 nm mit einer Wellenlängenkorrektur von 570 nm abgelesen.Plasmakonzentrationen von ICAM-1 (Probenverdünnung 1:1.000), SAA (Probenverdünnung 1:1.000) und IFN-γ (Probenverdünnung 1:2) wurden unter Anwendung der Multi-Array-Technologie von Meso Scale Discovery gemäß den Anweisungen des Herstellers gemessen. ICAM-1 und SAA wurden im Vascular Injury Panel (K15198D-1) gemessen, und IFN-γ wurde im Proinflammatory Panel (K15049D-1) nachgewiesen, beides gemäß den Anweisungen des Herstellers. Kurz gesagt, die Platten wurden dreimal mit 150 µl Waschpuffer pro Vertiefung gewaschen, und 25 µl verdünnte Probe, Kalibrator oder Kontrolle wurden pro Vertiefung (für ICAM-1 und SAA) oder 50 µl verdünnte Probe hinzugefügt. Kalibrator oder Kontrolle für IFN-γ. Die Platten wurden dann bei Raumtemperatur für 2 Stunden unter Schütteln inkubiert. Nach der Inkubation wurden die Platten dreimal mit 150 &mgr;l Waschpuffer pro Vertiefung gewaschen; 25 ul Detektionsantikörper wurden in jede Vertiefung gegeben; und die Platte wurde für eine weitere Stunde (für ICAM-1 und SAA) oder 2 Stunden für IFN-γ unter Schütteln bei Raumtemperatur inkubiert. Schließlich wurden die Platten dreimal mit 150 &mgr;l Waschpuffer pro Vertiefung gewaschen, und 150 &mgr;l 1 × Lesepuffer wurden pro Vertiefung (für ICAM-1 und SAA) oder 2 × Lesepuffer für IFN-&ggr; hinzugefügt. Signalintensitäten wurden mit einem MESO QuickPlex SQ 120-Instrument und der Discovery Workbench-Software, Version 4.0 (Meso Scale Discovery), erfasst und analysiert. Die durchschnittlichen Proteinkonzentrationen aus der oberen Hohlvene (SVC) und der unteren Hohlvene (IVC) und dem rechten Vorhof (RA) werden angegeben. die Platten wurden dreimal mit 150 &mgr;l Waschpuffer pro Vertiefung gewaschen, und 150 &mgr;l 1 × Lesepuffer wurden pro Vertiefung (für ICAM-1 und SAA) oder 2 × Lesepuffer für IFN-&ggr; hinzugefügt. Signalintensitäten wurden mit einem MESO QuickPlex SQ 120-Instrument und der Discovery Workbench-Software, Version 4.0 (Meso Scale Discovery), erfasst und analysiert. Die durchschnittlichen Proteinkonzentrationen aus der oberen Hohlvene (SVC) und der unteren Hohlvene (IVC) und dem rechten Vorhof (RA) werden angegeben. die Platten wurden dreimal mit 150 &mgr;l Waschpuffer pro Vertiefung gewaschen, und 150 &mgr;l 1 × Lesepuffer wurden pro Vertiefung (für ICAM-1 und SAA) oder 2 × Lesepuffer für IFN-&ggr; hinzugefügt. Signalintensitäten wurden mit einem MESO QuickPlex SQ 120-Instrument und der Discovery Workbench-Software, Version 4.0 (Meso Scale Discovery), erfasst und analysiert. Die durchschnittlichen Proteinkonzentrationen aus der oberen Hohlvene (SVC) und der unteren Hohlvene (IVC) und dem rechten Vorhof (RA) werden angegeben.Markierungsfreie quantitative EntdeckungsproteomikProbenvorbereitung für die ProteomikProtein wurde aus HUVECs und HUCMSCs extrahiert, und DNA wurde in 40 &mgr;l Lysepuffer (1 % SDS, 0,1 M ABC, 1,25 × PIC) in AFA-TUBE TPX Strips auf einem Covaris LE220Rsc durch fokussierte Ultraschallbehandlung (PIP 450 W, DF 25 %, CPB 200, zwei Wiederholungen, 300-Sekunden-Impuls, 20 °C). Die Proben wurden von Trümmern befreit (2.500 g für 5 Minuten) und das Protein quantifiziert (Pierce BCA, 23225). Proben von 30 &mgr;g Zellprotein wurden mit Lysepuffer auf 50 &mgr;l aufgefüllt, und 16,6 &mgr;l Reduktions- und Alkylierungspuffer (40 mM TCEP, 160 mM CAA, 200 mM ABC) wurden hinzugefügt. Sekretierte Proteine im CM (200 &mgr;l) wurden konzentriert (Gefriertrocknung über Nacht) und in 40 &mgr;l 10 mM TCEP und 40 mM CAA rekonstituiert. Zelluläre und sezernierte Proteine wurden unter Verwendung des SP3-Protokolls mit einstufiger Reduktion und Alkylierung 44 hergestellt auf einer Beckman Biomek i7 Workstation. Die Proben wurden für 5 Minuten bei 95°C inkubiert und auf Raumtemperatur abgekühlt. Proteine wurden an 250-μg-paramagnetische Kügelchen gebunden (1:1-Verhältnis von hydrophilen/hydrophoben Kügelchen) durch Zugabe von Acetonitril (ACN) zu 50 % für zelluläre Proteine bzw. 70 % für sezernierte Proteine. Die Proben wurden zweimal mit 80 % Ethanol und einmal mit 100 % ACN gewaschen, bevor sie in 35 &mgr;l 100 mM ABC rekonstituiert wurden. Der Verdau wurde über Nacht bei 37 °C unter Verwendung einer Trypsin/LysC-Enzymmischung (Promega) bei einem Verhältnis von Protein:Enzym von 50:1 für zelluläre Proteine bzw. 250 ng für sezernierte Proteine abgeschlossen. Die Reaktion wurde mit FA (0,1 %) gestoppt, und die Peptide wurden bis zur Analyse ohne weitere Konditionierung oder Reinigung bei −80 °C gelagert.Proteomanalyse durch datenunabhängige Akquisition LC–MSDie Menge des injizierten tryptischen Verdaus wurde auf 40 ng eingestellt, das verfügbare Material für die niedrigst konzentrierte Probe. Die Peptide wurden auf einer 25-cm-Aurora-Serie mit Emittersäule (CSI, 25 cm × 75 µm ID, 1,6 µm C18, IonOpticks, installiert in der Nano-Elektrospray-Quelle (CaptiveSpray-Quelle, Bruker Daltonics) bei 50 °C unter Verwendung von aufgelöst UltiMate 3000 (Thermo Fisher Scientific Dionex) gekoppelt mit TIMS-Quadrupol-Flugzeitinstrument (timsTOF Pro2, Bruker Daltonics) und gemessen im diaPASEF-Modus Die mobilen Phasen Wasser/0,1 % FA und ACN/0,1 % FA (A und B, jeweils) wurden in den linearen Gradienten angewendet, beginnend mit 2 % B und ansteigend auf 17 % in 87 Minuten, gefolgt von einer Erhöhung auf 25 % B in 93 Minuten, 37 % B in 98 Minuten und 80 % B in 99–104 Minuten, und die Säule wurde in den nächsten 15 Minuten in 2 % B äquilibriert.m / z [Th], 1/ K0 [Th]: 622,0289 , 0,9848; 922.0097, 1.1895; 1221.9906, 1.3820). Das diaPASEF-Fensterschema reichte in der Dimension m / z von 396 bis 1.103 Th und in der Dimension 1/ K 0 0,7–1,3 Vs cm-2, mit 59 × 12 Th-Fenstern). Alle Messungen wurden im Modus „Low Sample Amount“ mit einer Rampenzeit von 166 ms durchgeführt.Proteinidentifizierung und -quantifizierungDie Rohdaten wurden mit DIA-NN 1.8 (Ref. 45 ) mit dem Ionenmobilitätsmodul für diaPASEF verarbeitet (Demichev et al., www.biorxiv.org/content/10.1101/2021.03.08.434385v1 (2021)) . Die Massengenauigkeiten von MS2 und MS1 wurden beide auf 10 ppm eingestellt, und die Scanfenstergröße wurde auf 10 eingestellt. DIA-NN wurde im bibliotheksfreien Modus mit Standardeinstellungen (Fasta Digest und Deep-Learning-basierte Spektren, RT- und IMs-Vorhersage) ausgeführt. unter Verwendung der UniProt Human Reference Proteome Annotations ( UP000005640_9606 , heruntergeladen am 20. Dezember 2019) 46 und der Match-Between-Runs (MBR)-Option.LC-MS von Zellen und CMDie Proben wurden mit internen Standards bis zu einer Endkonzentration von 1,0 ng ml −1 versetzt und mittels Festphasenextraktion wie zuvor beschrieben präpariert 47 , und 200 &mgr;l CM-Proben wurden verwendet. Mediumproben wurden in 200 &mgr;l 40% MeOH und Zellpelletproben in 100 &mgr;l 40% MeOH rekonstituiert. Die LC-MS-Analyse wurde mit zwei LC-30AD-Pumpen, einem SIL-30AC-Autosampler und einem CTO-20AC-Säulenofen (alle Shimadzu) durchgeführt. Der Autosampler wurde bei 6 °C gehalten. Vierzig Mikroliter wurden injiziert, und die Trennung wurde auf einer Kinetex C18-Säule (Phenomenex, 50 × 2,1 mm, 1,7 &mgr;m) unter Verwendung eines Gradienten von 0,01 % Essigsäure (Fluka) in Wasser (Honeywell-Riedel de Haën; A) und 0,01 % Essigsäure in MeOH (
OMNIA TEMPUS HABENT
Diagnose IPAH im Februar 2013, in Behandlung bei OA Dr. Ulrich Krüger, jetzt Dr. Fischer Herzzentrum Duisburg, Medikamente: Sildenafil, Bosentan jetzt Macitentan, Subkutane Treprostinilpumpe, seit Januar 2024 getunnelter ZVK mit externer Pumpe (Groshongkatheter), 24/7 Sauerstoff, Marcumar, Diuretika
Bitte Anmelden oder Registrieren um der Konversation beizutreten.
27 Jun 2022 18:28 #1520
von danny
OMNIA TEMPUS HABENT
Diagnose IPAH im Februar 2013, in Behandlung bei OA Dr. Ulrich Krüger, jetzt Dr. Fischer Herzzentrum Duisburg, Medikamente: Sildenafil, Bosentan jetzt Macitentan, Subkutane Treprostinilpumpe, seit Januar 2024 getunnelter ZVK mit externer Pumpe (Groshongkatheter), 24/7 Sauerstoff, Marcumar, Diuretika
danny antwortete auf Aus mesenchymalen Stammzellen der menschlichen Nabelschnur gewonnene Behandlung
FAZ Artikel zum Thema
www.faz.net/aktuell/wissen/medizin-ernae...-leben-18099979.html
www.faz.net/aktuell/wissen/medizin-ernae...-leben-18099979.html
OMNIA TEMPUS HABENT
Diagnose IPAH im Februar 2013, in Behandlung bei OA Dr. Ulrich Krüger, jetzt Dr. Fischer Herzzentrum Duisburg, Medikamente: Sildenafil, Bosentan jetzt Macitentan, Subkutane Treprostinilpumpe, seit Januar 2024 getunnelter ZVK mit externer Pumpe (Groshongkatheter), 24/7 Sauerstoff, Marcumar, Diuretika
Bitte Anmelden oder Registrieren um der Konversation beizutreten.
